Heute sind wir in der Wissenschaft und Technologie, wo wir Menschen das, was die Natur bereits geleistet hat, verdoppeln und dann verbessern können. Auch wir können das Anorganische in das Organische verwandeln. Auch wir können Genome lesen und interpretieren, aber auch modifizieren. Und auch wir können genetische Vielfalt schaffen, indem wir die beträchtliche Summe dessen, was die Natur bereits hervorgebracht hat, ergänzen.
~
Dr. George Church
Exzerpt aus dem Buch „Storing Digital Binary Data in Cellular DNA: The New Paradigm“ (2020)
p.24
Alle Religionen glauben an Wunder, aber sie kommen zu zufälligennZeiten, und oft nur nachh einem echten Gebet. Gott mit den menschlichen Genen zu spielen ist die Mutter aller Wunder. Das Schneiden undd Einfügen vonnDNA ist einnwahrer "game changer" in der Genrevolution. Die Menschen, die heute in der Genetik forschen, zählen sich zu den Glücklichen. Sie zelebrieren eine neue Technik, die in jeder Hinsicht revolutionär ist.
All religions believe in miracles, but they come at random times, and often only after a genuine prayer.Playing God with human genes is the mother of all miracles. Editingdcutting and pastingdDNA is atrue “game changer” in the gene revolution. The people who are doing research in genetics today countthemselves among the lucky ones. They are celebrating a new technique that is, by all counts,revolutionary. The cause of their uncharacteristic giddiness is a remarkably reliable method of editingthe human genome.
https://www.youtube.com/watch?v=60Gi5lqL-dA
Khan, F., Ncube, C., Ramasamy, L. K., Kadry, S., & Nam, Y. (2020). A Digital DNA Sequencing Engine for Ransomware Detection Using Machine Learning.
IEEE Access. https://doi.org/10.1109/ACCESS.2020.3003785
Wagner, R., Haefner, B., Biehler, M., & Lanza, G. (2020). Digital DNA in quality control cycles of high-precision products.
CIRP Annals. https://doi.org/10.1016/j.cirp.2020.03.020
Meiser, L. C., Antkowiak, P. L., Koch, J., Chen, W. D., Kohll, A. X., Stark, W. J., Heckel, R., & Grass, R. N. (2020). Reading and writing digital data in DNA.
Nature Protocols. https://doi.org/10.1038/s41596-019-0244-5
Digital DNA. (2020). In
Chemistry and Industry (London). https://doi.org/10.1002/cind.8411_7.x
Wöhrle, J., Krämer, S. D., Meyer, P. A., Rath, C., Hügle, M., Urban, G. A., & Roth, G. (2020). Digital DNA microarray generation on glass substrates.
Scientific Reports. https://doi.org/10.1038/s41598-020-62404-1
Cresci, S., Pietro, R. Di, Petrocchi, M., Spognardi, A., & Tesconi, M. (2018). Social Fingerprinting: Detection of Spambot Groups Through DNA-Inspired Behavioral Modeling.
IEEE Transactions on Dependable and Secure Computing. https://doi.org/10.1109/TDSC.2017.2681672
Hood, L., & Galas, D. (2003). The digital code of DNA. In
Nature. https://doi.org/10.1038/nature01410
Ping, Z., Ma, D., Huang, X., Chen, S., Liu, L., Guo, F., Zhu, S. J., & Shen, Y. (2019). Carbon-based archiving: Current progress and future prospects of DNA-based data storage. In
GigaScience. https://doi.org/10.1093/gigascience/giz075
Lee, W., Chen, Q., Fan, X., & Yoon, D. K. (2016). Digital DNA detection based on a compact optofluidic laser with ultra-low sample consumption.
Lab on a Chip. https://doi.org/10.1039/c6lc01258b
Raymond, T., Li, V., & Argyriou, V. (2020). Growth-based 3D modelling using stem-voxels encoded in digital-DNA structures.
SIGGRAPH Asia 2020 Posters. SA 2020. https://doi.org/10.1145/3415264.3425443
Panda, D., Molla, K. A., Baig, M. J., Swain, A., Behera, D., & Dash, M. (2018). DNA as a digital information storage device: hope or hype? In
3 Biotech. https://doi.org/10.1007/s13205-018-1246-7
Riojas, M. A., McGough, K. J., Rider-Riojas, C. J., Rastogi, N., & Hazbón, M. H. (2018). Phylogenomic analysis of the species of the mycobacterium tuberculosis complex demonstrates that mycobacterium africanum, mycobacterium bovis, mycobacterium caprae, mycobacterium microti and mycobacterium pinnipedii are later heterotypic synonyms of mycobacterium tuberculosis.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.002507
Sun, K., Jiang, P., & Chan, K. A. (2015). The impact of digital DNA counting technologies on noninvasive prenatal testing.
Expert Review of Molecular Diagnostics. https://doi.org/10.1586/14737159.2015.1084227
Qi, Y., Zhai, Y., Fan, W., Ren, W., Li, Z., & Liu, C. (2021). Click chemistry-actuated digital DNA walker confined on a single particle toward absolute MicroRNA quantification.
Analytical Chemistry. https://doi.org/10.1021/acs.analchem.0c04073
Yan, Y. H., Zhang, D. Y., & Wu, L. R. (2021). Encoding multiple digital DNA signals in a single analog channel.
Nucleic Acids Research. https://doi.org/10.1093/NAR/GKAA303
Gevensleben, H., Garcia-Murillas, I., Graeser, M. K., Schiavon, G., Osin, P., Parton, M., Smith, I. E., Ashworth, A., & Turner, N. C. (2013). Noninvasive detection of HER2 amplification with plasma DNA digital PCR.
Clinical Cancer Research. https://doi.org/10.1158/1078-0432.CCR-12-3768
Wang, F., Lv, H., Li, Q., Li, J., Zhang, X., Shi, J., Wang, L., & Fan, C. (2020). Implementing digital computing with DNA-based switching circuits.
Nature Communications. https://doi.org/10.1038/s41467-019-13980-y
Kint, S., De Spiegelaere, W., De Kesel, J., Vandekerckhove, L., & Van Criekinge, W. (2018). Evaluation of bisulfite kits for DNA methylation profiling in terms of DNA fragmentation and DNA recovery using digital PCR.
PLoS ONE. https://doi.org/10.1371/journal.pone.0199091
George, A. K., Kunnummal, I. O., Alazzawi, L., & Singh, H. (2020). Design of DNA Digital Circuits.
IEEE Potentials. https://doi.org/10.1109/MPOT.2018.2859443
Lee, H. H., Kalhor, R., Goela, N., Bolot, J., & Church, G. M. (2019). Terminator-free template-independent enzymatic DNA synthesis for digital information storage.
Nature Communications. https://doi.org/10.1038/s41467-019-10258-1
Björkesten, J., Patil, S., Fredolini, C., Lönn, P., & Landegren, U. (2020). A multiplex platform for digital measurement of circular DNA reaction products.
Nucleic Acids Research. https://doi.org/10.1093/nar/gkaa419
Badawi, H. F., Laamarti, F., & Saddik, A. El. (2021). Devising digital twins DNA paradigm for modeling ISO-based city services.
Sensors (Switzerland). https://doi.org/10.3390/s21041047
Lee, H. H., Kalhor, R., Goela, N., Bolot, J., & Church, G. M. (2018). Enzymatic DNA synthesis for digital information storage.
BioRxiv. https://doi.org/10.1101/348987
Podbiel, D., Laermer, F., Zengerle, R., & Hoffmann, J. (2020). Fusing MEMS technology with lab-on-chip: nanoliter-scale silicon microcavity arrays for digital DNA quantification and multiplex testing.
Microsystems and Nanoengineering. https://doi.org/10.1038/s41378-020-00187-1
Ledenyov, D. O., & Ledenyov, V. O. (2016). Digital DNA of Economy of Scale and Scope.
SSRN Electronic Journal. https://doi.org/10.2139/ssrn.2718931
Weisenberger, D. J., Trinh, B. N., Campan, M., Sharma, S., Long, T. I., Ananthnarayan, S., Liang, G., Esteva, F. J., Hortobagyi, G. N., McCormick, F., Jones, P. A., & Laird, P. W. (2008). DNA methylation analysis by digital bisulfite genomic sequencing and digital MethyLight.
Nucleic Acids Research. https://doi.org/10.1093/nar/gkn455
Sinha, M., Mack, H., Coleman, T. P., & Fraley, S. I. (2018). A High-Resolution Digital DNA Melting Platform for Robust Sequence Profiling and Enhanced Genotype Discrimination.
SLAS Technology. https://doi.org/10.1177/2472630318769846
Akram, F., Haq, I. ul, Ali, H., & Laghari, A. T. (2018). Trends to store digital data in DNA: an overview. In
Molecular Biology Reports. https://doi.org/10.1007/s11033-018-4280-y
Kühnemund, M., Hernández-Neuta, I., Sharif, M. I., Cornaglia, M., Gijs, M. A. M., & Nilsson, M. (2017). Sensitive and inexpensive digital DNA analysis by microfluidic enrichment of rolling circle amplified single-molecules.
Nucleic Acids Research. https://doi.org/10.1093/nar/gkw1324
Aliyu, H., Lebre, P., Blom, J., Cowan, D., & De Maayer, P. (2016). Phylogenomic re-assessment of the thermophilic genus Geobacillus.
Systematic and Applied Microbiology. https://doi.org/10.1016/j.syapm.2016.09.004
George, A. K., & Singh, H. (2016). Enzyme-free scalable DNA digital design techniques: A review. In
IEEE Transactions on Nanobioscience. https://doi.org/10.1109/TNB.2016.2623218
Parshina, S. N., Strepis, N., Aalvink, S., Nozhevnikova, A. N., Stams, A. J. M., & Sousa, D. Z. (2019). Trichococcus shcherbakoviae sp. nov., isolated from a laboratory-scale anaerobic EGSB bioreactor operated at low temperature.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.003193
Sun, L., He, J., Luo, J., & Coy, D. H. (2019). DNA and the Digital Data Storage.
Health Sci J.
Grass, R. N., Heckel, R., Dessimoz, C., & Stark, W. J. (2020). Genomic Encryption of Digital Data Stored in Synthetic DNA.
Angewandte Chemie - International Edition. https://doi.org/10.1002/anie.202001162
Arter, W. E., Yusim, Y., Peter, Q., Taylor, C. G., Klenerman, D., Keyser, U. F., & Knowles, T. P. J. (2020). Digital Sensing and Molecular Computation by an Enzyme-Free DNA Circuit.
ACS Nano. https://doi.org/10.1021/acsnano.0c00628
Auch, A. F., von Jan, M., Klenk, H. P., & Göker, M. (2010). Digital DNA-DNA hybridization for microbial species delineation by means of genome-to-genome sequence comparison.
Standards in Genomic Sciences. https://doi.org/10.4056/sigs.531120
Manor, I. (2019). Digital DNA: disruption and the challenges for global governance.
Cambridge Review of International Affairs. https://doi.org/10.1080/09557571.2018.1563971
Orata, F. D., Meier-Kolthoff, J. P., Sauvageau, D., & Stein, L. Y. (2018). Phylogenomic analysis of the gammaproteobacterial methanotrophs (order methylococcales) calls for the reclassification of members at the genus and species levels.
Frontiers in Microbiology. https://doi.org/10.3389/fmicb.2018.03162
Lee, H., Wiegand, D. J., Griswold, K., Punthambaker, S., Chun, H., Kohman, R. E., & Church, G. M. (2020). Photon-directed multiplexed enzymatic DNA synthesis for molecular digital data storage.
Nature Communications. https://doi.org/10.1038/s41467-020-18681-5
Goldman, N., Hesketh, E. E., & Sayir, J. (2018). Improving communication for interdisciplinary teams working on storage of digital information in DNA.
F1000Research. https://doi.org/10.12688/f1000research.13482.1
Grass, R. N., Heckel, R., Dessimoz, C., & Stark, W. J. (2020). Genomic Encryption of Digital Data Stored in Synthetic DNA.
Angewandte Chemie. https://doi.org/10.1002/ange.202001162
Chen, K., Kong, J., Zhu, J., Ermann, N., Predki, P., & Keyser, U. F. (2019). Digital Data Storage Using DNA Nanostructures and Solid-State Nanopores.
Nano Letters. https://doi.org/10.1021/acs.nanolett.8b04715
Ceze, L., Nivala, J., & Strauss, K. (2019). Molecular digital data storage using DNA. In
Nature Reviews Genetics. https://doi.org/10.1038/s41576-019-0125-3
Cresci, S., Petrocchi, M., Spognardi, A., & Tognazzi, S. (2019). On the capability of evolved spambots to evade detection via genetic engineering.
Online Social Networks and Media. https://doi.org/10.1016/j.osnem.2018.10.005
Trotsyuk, R., & Santos, V. (2019). The enterprise DNA: Static and dynamic digital representation of organizations.
International Journal of Engineering and Advanced Technology. https://doi.org/10.35940/ijeat.F9544.088619
Jin, H., Wang, H., Zhang, Y., Hu, T., Lin, Z., Liu, B., Ma, J., Wang, X., Liu, Q., Lin, X., & Xie, Z. (2020). Description of azotobacter chroococcum subsp. Isscasi subsp. nov. isolated from paddy soil and establishment of azotobacter chroococcum subsp. chroococcum subsp. nov.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004026
Sombolestani, A. S., Cleenwerck, I., Cnockaert, M., Borremans, W., Wieme, A. D., Moutia, Y., Spaepen, S., De Vuyst, L., & Vandamme, P. (2021). Gluconacetobacter dulcium sp. Nov., a novel gluconacetobacter species from sugar-rich environments.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004569
Zhou, L. Y., Meng, X., Zhong, Y. L., Li, G. Y., Du, Z. J., & Mu, D. S. (2020). Dokdonia sinensis sp. Nov., a flavobacterium isolated from surface seawater.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.003949
Liu, Q., Xin, Y. H., Chen, X. L., Liu, H. C., Zhou, Y. G., & Chen, W. X. (2018). Arthrobacter ruber sp. Nov., isolated from glacier ice.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.002719
Grass, R. N., Heckel, R., Puddu, M., Paunescu, D., & Stark, W. J. (2015). Robust chemical preservation of digital information on DNA in silica with error-correcting codes.
Angewandte Chemie - International Edition. https://doi.org/10.1002/anie.201411378
Lin, S. Y., Chen, W. M., Huang, G. H., Hameed, A., Chang, C. T., Tsai, C. F., & Young, C. C. (2020). Flavobacterium supellecticarium sp. Nov., isolated from an abandoned construction timber.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004227
Cevallos, Y., Tello-Oquendo, L., Inca, D., Samaniego, N., Santillán, I., Shirazi, A. Z., & Gomez, G. A. (2020). On the efficient digital code representation in DNA-based data storage.
Proceedings of the 7th ACM International Conference on Nanoscale Computing and Communication, NanoCom 2020. https://doi.org/10.1145/3411295.3411314
Wang, Y., Zhang, Y., & Zhao, Y. (2019). DNA digital data storage based on distributed method.
ACM International Conference Proceeding Series. https://doi.org/10.1145/3340074.3340082
Zhang, Z., Zhao, S., Hu, F., Yang, G., Li, J., Tian, H., & Peng, N. (2020). An LED-Driven AuNPs-PDMS microfluidic chip and integrated device for the detection of digital loop-mediated isothermal DNA amplification.
Micromachines. https://doi.org/10.3390/mi11020177
Hoshino, T., & Inagaki, F. (2012). Molecular quantification of environmental DNA using microfluidics and digital PCR.
Systematic and Applied Microbiology. https://doi.org/10.1016/j.syapm.2012.06.006
Garcia-Murillas, I., Lambros, M., & Turner, N. C. (2013). Determination of HER2 amplification status on tumour DNA by digital PCR.
PLoS ONE. https://doi.org/10.1371/journal.pone.0083409
Koch, J., Gantenbein, S., Masania, K., Stark, W. J., Erlich, Y., & Grass, R. N. (2020). A DNA-of-things storage architecture to create materials with embedded memory. In
Nature Biotechnology. https://doi.org/10.1038/s41587-019-0356-z
Pasricha, N., & Hayes, C. (2019). Detecting bot behaviour in social media using digital DNA compression.
CEUR Workshop Proceedings.
Madhaiyan, M., Wirth, J. S., & Saravanan, V. S. (2020). Phylogenomic analyses of the staphylococcaceae family suggest the reclassification of five species within the genus staphylococcus as heterotypic synonyms, the promotion of five subspecies to novel species, the taxonomic reassignment of five staphylococcus species to mammaliicoccus gen. Nov., and the formal assignment of nosocomiicoccus to the family staphylococcaceae.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004498
O’Hara, R., Tedone, E., Ludlow, A., Huang, E., Arosio, B., Mari, D., & Shay, J. W. (2019). Quantitative mitochondrial DNA copy number determination using droplet digital PCR with single-cell resolution.
Genome Research. https://doi.org/10.1101/gr.250480.119
Rondelez, Y., & Gines, G. (2020). Multiplex Digital MicroRNA Detection Using Cross-Inhibitory DNA Circuits.
ACS Sensors. https://doi.org/10.1021/acssensors.0c00593
Lin, S. Y., Hameed, A., Huang, H. I., & Young, C. C. (2020). Allorhizobium terrae sp. Nov., isolated from paddy soil, and reclassification of rhizobium oryziradicis (zhao et al. 2017) as allorhizobium oryziradicis comb. nov.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.003770
Qian, L., & Winfree, E. (2011). Scaling up digital circuit computation with DNA strand displacement cascades.
Science. https://doi.org/10.1126/science.1200520
Zhang, S., Huang, B., Song, X., Zhang, T., Wang, H., & Liu, Y. (2019). A high storage density strategy for digital information based on synthetic DNA.
3 Biotech. https://doi.org/10.1007/s13205-019-1868-4
Pavšič, J., Žel, J., & Milavec, M. (2016). Assessment of the real-time PCR and different digital PCR platforms for DNA quantification.
Analytical and Bioanalytical Chemistry. https://doi.org/10.1007/s00216-015-9107-2
Han, J., Lee, J. Y., & Bae, Y. K. (2019). Application of digital PCR for assessing DNA fragmentation in cytotoxicity response.
Biochimica et Biophysica Acta - General Subjects. https://doi.org/10.1016/j.bbagen.2019.05.001
Yukl, S. A., Kaiser, P., Kim, P., Li, P., & Wong, J. K. (2014). Advantages of using the QIAshredder instead of restriction digestion to prepare DNA for droplet digital PCR.
BioTechniques. https://doi.org/10.2144/000114159
Strain, M. C., Lada, S. M., Luong, T., Rought, S. E., Gianella, S., Terry, V. H., Spina, C. A., Woelk, C. H., & Richman, D. D. (2013). Highly Precise Measurement of HIV DNA by Droplet Digital PCR.
PLoS ONE. https://doi.org/10.1371/journal.pone.0055943
Silva, F. D. J., Ferreira, L. C., Campos, V. P., Cruz-Magalhães, V., Barros, A. F., Andrade, J. P., Roberts, D. P., De Souza, J. T., & Ochman, H. (2019). Complete Genome Sequence of the Biocontrol Agent Bacillus velezensis UFLA258 and Its Comparison with Related Species: Diversity within the Commons.
Genome Biology and Evolution. https://doi.org/10.1093/gbe/evz208
Marks, R. A., Vieira, D. K. S., Guterres, M. V., Oliveira, P. A. C., Fonte Boa, M. C. O., & Vilela Neto, O. P. (2021). Design and Test of Digital Logic DNA Systems.
IEEE Design and Test. https://doi.org/10.1109/MDAT.2021.3069369
Church, G. M., Gao, Y., & Kosuri, S. (2012). Next-generation digital information storage in DNA. In
Science. https://doi.org/10.1126/science.1226355
De Silva, P. Y., & Ganegoda, G. U. (2016). New Trends of Digital Data Storage in DNA. In
BioMed Research International. https://doi.org/10.1155/2016/8072463
Dong, L., Yoo, H. B., Wang, J., & Park, S. R. (2016). Accurate quantification of supercoiled DNA by digital PCR.
Scientific Reports. https://doi.org/10.1038/srep24230
Fries, R., & Durstewitz, G. (2001). Digital DNA signatures for animal tagging[2]. In
Nature Biotechnology. https://doi.org/10.1038/89213
Ahn, T., Ban, H., & Park, H. (2018). Storing Digital Information in Long-Read DNA.
Genomics & Informatics. https://doi.org/10.5808/gi.2018.16.4.e30
Guest, M. (2014). Building your digital DNA Lessons from digital leaders Contents The digital organisation.
Deloitte.
Chaudhry, V., Baindara, P., Pal, V. K., Chawla, N., Patil, P. B., & Korpole, S. (2016). Methylobacterium indicum sp. nov., a facultative methylotrophic bacterium isolated from rice seed.
Systematic and Applied Microbiology. https://doi.org/10.1016/j.syapm.2015.12.006
Gassa, A., Fassunke, J., Schueten, S., Kuhlmann, L., Scherer, M., Qien, J., Zhao, Y., Michel, M., Loeser, H., Wolf, J., Buettner, R., Doerr, F., Heldwein, M., Hagmeyer, L., Frank, K., Merkelbach-Bruse, S., Quaas, A., Bruns, C., Hekmat, K., … Alakus, H. (2020). Detection of circulating tumor DNA by digital droplet PCR in resectable lung cancer as a predictive tool for recurrence.
Lung Cancer. https://doi.org/10.1016/j.lungcan.2020.10.019
Cho, S. M., Shin, S., Kim, Y., Song, W., Hong, S. G., Jeong, S. H., Kang, M. S., & Lee, K. A. (2020). A novel approach for tuberculosis diagnosis using exosomal DNA and droplet digital PCR.
Clinical Microbiology and Infection. https://doi.org/10.1016/j.cmi.2019.11.012
Pinheiro, L. B., Coleman, V. A., Hindson, C. M., Herrmann, J., Hindson, B. J., Bhat, S., & Emslie, K. R. (2012). Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification.
Analytical Chemistry. https://doi.org/10.1021/ac202578x
Nell, R. J., van Steenderen, D., Menger, N. V., Weitering, T. J., Versluis, M., & van der Velden, P. A. (2020). Quantification of DNA methylation independent of sodium bisulfite conversion using methylation-sensitive restriction enzymes and digital PCR.
Human Mutation. https://doi.org/10.1002/humu.24111
Hindson, B. J., Ness, K. D., Masquelier, D. A., Belgrader, P., Heredia, N. J., Makarewicz, A. J., Bright, I. J., Lucero, M. Y., Hiddessen, A. L., Legler, T. C., Kitano, T. K., Hodel, M. R., Petersen, J. F., Wyatt, P. W., Steenblock, E. R., Shah, P. H., Bousse, L. J., Troup, C. B., Mellen, J. C., … Colston, B. W. (2011). High-throughput droplet digital PCR system for absolute quantitation of DNA copy number.
Analytical Chemistry. https://doi.org/10.1021/ac202028g
Laddha, R., & Honwadkar, K. (2016). Digital Data Storage on DNA.
International Journal of Computer Applications. https://doi.org/10.5120/ijca2016909700
Bagley, M. A. (2016). Digital DNA: The Nagoya Protocol, Intellectual Property Treaties, and Synthetic Biology.
SSRN Electronic Journal. https://doi.org/10.2139/ssrn.2725986
Krumbholz, M., Goerlitz, K., Albert, C., Lawlor, J., Suttorp, M., & Metzler, M. (2019). Large amplicon droplet digital PCR for DNA-based monitoring of pediatric chronic myeloid leukaemia.
Journal of Cellular and Molecular Medicine. https://doi.org/10.1111/jcmm.14321
Dallaway, E. (2008). Digital DNA.
Infosecurity. https://doi.org/10.1016/s1754-4548(08)70075-5
Church, G. M., Gao, Y., & Kosuri, S. (2012). Supplementary Materials for Next-Generation digital information storage in DNA.
Science (New York, N.Y.).
Ishii, H., Azuma, K., Sakai, K., Kawahara, A., Yamada, K., Tokito, T., Okamoto, I., Nishio, K., & Hoshino, T. (2015). Digital PCR analysis of plasma cell-free DNA for non-invasive detection of drug resistance mechanisms in EGFR mutant NSCLC: Correlation with paired tumor samples.
Oncotarget. https://doi.org/10.18632/oncotarget.5068
V, K. (2021). DNA; Digital Data Storage Device.
International Research Journal on Advanced Science Hub. https://doi.org/10.47392/irjash.2021.035
Carmean, D., Ceze, L., Seelig, G., Stewart, K., Strauss, K., & Willsey, M. (2019). DNA Data Storage and Hybrid Molecular-Electronic Computing.
Proceedings of the IEEE. https://doi.org/10.1109/JPROC.2018.2875386
Zou, Z., Guo, L., Ahmadi, P., Hartjen, P., Gosau, M., Smeets, R., & Kluwe, L. (2021). Two simple and inexpensive methods for preparing DNA suitable for digital PCR from a small number of cells in 96-well plates.
Journal of Clinical Laboratory Analysis. https://doi.org/10.1002/jcla.23513
Kohll, A. X., Antkowiak, P. L., Chen, W. D., Nguyen, B. H., Stark, W. J., Ceze, L., Strauss, K., & Grass, R. N. (2020). Stabilizing synthetic DNA for long-Term data storage with earth alkaline salts.
Chemical Communications. https://doi.org/10.1039/d0cc00222d
Thompson, D., Cognat, V., Goodfellow, M., Koechler, S., Heintz, D., Carapito, C., Van Dorsselaer, A., Mahmoud, H., Sangal, V., & Ismail, W. (2020). Phylogenomic Classification and Biosynthetic Potential of the Fossil Fuel-Biodesulfurizing Rhodococcus Strain IGTS8.
Frontiers in Microbiology. https://doi.org/10.3389/fmicb.2020.01417
Phadke, S. (2021). FinTech Future: The Digital DNA of Finance. In
FinTech Future: The Digital DNA of Finance. https://doi.org/10.4135/9789353885687
Perez-Toralla, K., Pereiro, I., Garrigou, S., Di Federico, F., Proudhon, C., Bidard, F. C., Viovy, J. L., Taly, V., & Descroix, S. (2019). Microfluidic extraction and digital quantification of circulating cell-free DNA from serum.
Sensors and Actuators, B: Chemical. https://doi.org/10.1016/j.snb.2019.01.159
O’Leary, B., Hrebien, S., Beaney, M., Fribbens, C., Garcia-Murillas, I., Jiang, J., Li, Y., Bartlett, C. H., André, F., Loibl, S., Loi, S., Cristofanilli, M., & Turner, N. C. (2019). Comparison of beaming and droplet digital PCR for circulating tumor DNA analysis.
Clinical Chemistry. https://doi.org/10.1373/clinchem.2019.305805
Baume, M., Cariou, A., Leveau, A., Fessy, N., Pastori, F., Jarraud, S., & Pierre, S. (2019). Quantification of Legionella DNA certified reference material by digital droplet PCR.
Journal of Microbiological Methods. https://doi.org/10.1016/j.mimet.2018.12.019
Rodriguez-Manzano, J., Karymov, M. A., Begolo, S., Selck, D. A., Zhukov, D. V., Jue, E., & Ismagilov, R. F. (2016). Reading Out Single-Molecule Digital RNA and DNA Isothermal Amplification in Nanoliter Volumes with Unmodified Camera Phones.
ACS Nano. https://doi.org/10.1021/acsnano.5b07338
Li, M., Chen, W. D., Papadopoulos, N., Goodman, S. N., Bjerregaard, N. C., Laurberg, S., Levin, B., Juhl, H., Arber, N., Moinova, H., Durkee, K., Schmidt, K., He, Y., Diehl, F., Velculescu, V. E., Zhou, S., Diaz, L. A., Kinzler, K. W., Markowitz, S. D., & Vogelstein, B. (2009). Sensitive digital quantification of DNA methylation in clinical samples.
Nature Biotechnology. https://doi.org/10.1038/nbt.1559
Newman, S., Stephenson, A. P., Willsey, M., Nguyen, B. H., Takahashi, C. N., Strauss, K., & Ceze, L. (2019). High density DNA data storage library via dehydration with digital microfluidic retrieval.
Nature Communications. https://doi.org/10.1038/s41467-019-09517-y
Hayashi Sant’Anna, F., Bach, E., Porto, R. Z., Guella, F., Hayashi Sant’Anna, E., & Passaglia, L. M. P. (2019). Genomic metrics made easy: what to do and where to go in the new era of bacterial taxonomy. In
Critical Reviews in Microbiology. https://doi.org/10.1080/1040841X.2019.1569587
Wambui, J., Cernela, N., Stevens, M. J. A., & Stephan, R. (2020). Draft Genome Sequence of Clostridium estertheticum CEST001, Belonging to a Novel Subspecies of C. estertheticum , Isolated from Chilled Vacuum-Packed Lamb Meat Imported to Switzerland .
Microbiology Resource Announcements. https://doi.org/10.1128/mra.00806-20
Servick, K. (2016). Rise of digital DNA raises biopiracy fears.
Science. https://doi.org/10.1126/science.aal0395
Khilko, Y., Weyman, P. D., Glass, J. I., Adams, M. D., McNeil, M. A., & Griffin, P. B. (2018). DNA assembly with error correction on a droplet digital microfluidics platform.
BMC Biotechnology. https://doi.org/10.1186/s12896-018-0439-9
Husale, S., Persson, H. H. J., & Sahin, O. (2009). DNA nanomechanics allows direct digital detection of complementary DNA and microRNA targets.
Nature. https://doi.org/10.1038/nature08626
Batrakou, D. G., Heron, E. D., & Nieduszynski, C. A. (2018). Rapid high-resolution measurement of DNA replication timing by droplet digital PCR.
Nucleic Acids Research. https://doi.org/10.1093/nar/gky590
O’Hara, R., Tedone, E., Ludlow, A., Huang, E., Arosio, B., Mari, D., & Shay, J. (2019). Quantitative mitochondrial DNA copy number determination using droplet digital PCR with single cell resolution: a focus on aging and cancer.
BioRxiv. https://doi.org/10.1101/579789
O’Keefe, C. M., Giammanco, D., Li, S., Pisanic, T. R., & Wang, T. H. J. (2019). Multilayer microfluidic array for highly efficient sample loading and digital melt analysis of DNA methylation.
Lab on a Chip. https://doi.org/10.1039/c8lc01189c
Takahashi, Y. (2019). Consumer behavior DNA for realizing flexible digital marketing.
Fujitsu Scientific and Technical Journal.
Demeke, T., Eng, M., Holigroski, M., & Lee, S. J. (2021). Effect of Amount of DNA on Digital PCR Assessment of Genetically Engineered Canola and Soybean Events.
Food Analytical Methods. https://doi.org/10.1007/s12161-020-01889-y
Sanders, R., Huggett, J. F., Bushell, C. A., Cowen, S., Scott, D. J., & Foy, C. A. (2011). Evaluation of digital PCR for absolute DNA quantification.
Analytical Chemistry. https://doi.org/10.1021/ac103230c
Hrebien, S., O’Leary, B., Beaney, M., Schiavon, G., Fribbens, C., Bhambra, A., Johnson, R., Garcia-Murillas, I., & Turner, N. (2016). Reproducibility of digital PCR assays for circulating tumor DNA analysis in advanced breast cancer.
PLoS ONE. https://doi.org/10.1371/journal.pone.0165023
Garafutdinov, R. R., Sakhabutdinova, A. R., Slominsky, P. A., Aminev, F. G., & Chemeris, A. V. (2020). A new digital approach to SNP encoding for DNA identification.
Forensic Science International. https://doi.org/10.1016/j.forsciint.2020.110520
Alcaide, M., Cheung, M., Hillman, J., Rassekh, S. R., Deyell, R. J., Batist, G., Karsan, A., Wyatt, A. W., Johnson, N., Scott, D. W., & Morin, R. D. (2020). Evaluating the quantity, quality and size distribution of cell-free DNA by multiplex droplet digital PCR.
Scientific Reports. https://doi.org/10.1038/s41598-020-69432-x
Ma, L., Wagner, J., Rice, J. J., Hu, W., Levine, A. J., & Stolovitzky, G. A. (2005). A plausible model for the digital response of p53 to DNA damage.
Proceedings of the National Academy of Sciences of the United States of America. https://doi.org/10.1073/pnas.0501352102
Kojabad, A. A., Farzanehpour, M., Galeh, H. E. G., Dorostkar, R., Jafarpour, A., Bolandian, M., & Nodooshan, M. M. (2021). Droplet digital PCR of viral DNA/RNA, current progress, challenges, and future perspectives. In
Journal of Medical Virology. https://doi.org/10.1002/jmv.26846
Sefrioui, D., Sarafan-Vasseur, N., Beaussire, L., Baretti, M., Gangloff, A., Blanchard, F., Clatot, F., Sabourin, J. C., Sesboüé, R., Frebourg, T., Michel, P., & Di Fiore, F. (2015). Clinical value of chip-based digital-PCR platform for the detection of circulating DNA in metastatic colorectal cancer.
Digestive and Liver Disease. https://doi.org/10.1016/j.dld.2015.05.023
Zou, F., Ruan, Q., Lin, X., Zhang, M., Song, Y., Zhou, L., Zhu, Z., Lin, S., Wang, W., & Yang, C. J. (2019). Rapid, real-time chemiluminescent detection of DNA mutation based on digital microfluidics and pyrosequencing.
Biosensors and Bioelectronics. https://doi.org/10.1016/j.bios.2018.09.092
Kühnemund, M., & Nilsson, M. (2015). Digital quantification of rolling circle amplified single DNA molecules in a resistive pulse sensing nanopore.
Biosensors and Bioelectronics. https://doi.org/10.1016/j.bios.2014.06.040
Lu, B., Zeng, F., Xing, W., Liang, L., Huo, J., Tan, C., Zhu, L., & Liu, Z. (2020). Decreased mitochondrial DNA copy number in children with cerebral palsy quantified by droplet digital PCR.
Clinica Chimica Acta. https://doi.org/10.1016/j.cca.2020.01.018
Luna, F. M. F., Tsui, N. B. Y., Chan, K. C. A., Leung, T. Y., Lau, T. K., Charoenkwan, P., Chow, K. C. K., Lo, W. Y. W., Wanapirak, C., Sanguansermsri, T., Cantor, C. R., Chiu, R. W. K., & Lo, Y. M. D. (2008). Noninvasive prenatal diagnosis of monogenic diseases by digital size selection and relative mutation dosage on DNA in maternal plasma.
Proceedings of the National Academy of Sciences of the United States of America. https://doi.org/10.1073/pnas.0810373105
Blawat, M., Gaedke, K., Hütter, I., Chen, X. M., Turczyk, B., Inverso, S., Pruitt, B. W., & Church, G. M. (2016). Forward error correction for DNA data storage.
Procedia Computer Science. https://doi.org/10.1016/j.procs.2016.05.398
Lanman, R. B., Mortimer, S. A., Zill, O. A., Sebisanovic, D., Lopez, R., Blau, S., Collisson, E. A., Divers, S. G., Hoon, D. S. B., Scott Kopetz, E., Lee, J., Nikolinakos, P. G., Baca, A. M., Kermani, B. G., Eltoukhy, H., & Talasaz, A. A. (2015). Analytical and clinical validation of a digital sequencing panel for quantitative, highly accurate evaluation of cell-free circulating tumor DNA.
PLoS ONE. https://doi.org/10.1371/journal.pone.0140712
Akkasaligar, P. T., & Biradar, S. (2020). Selective medical image encryption using DNA cryptography. In
Information Security Journal. https://doi.org/10.1080/19393555.2020.1718248
Gansen, A., Herrick, A. M., Dimov, I. K., Lee, L. P., & Chiu, D. T. (2012). Digital LAMP in a sample self-digitization (SD) chip.
Lab on a Chip. https://doi.org/10.1039/c2lc21247a
Lee, J. S., Kim, M., Seong, M. W., Kim, H. S., Lee, Y. K., & Kang, H. J. (2020). Plasma vs. serum in circulating tumor DNA measurement: Characterization by DNA fragment sizing and digital droplet polymerase chain reaction.
Clinical Chemistry and Laboratory Medicine. https://doi.org/10.1515/cclm-2019-0896
Langouche, L., Aralar, A., Sinha, M., Lawrence, S. M., Fraley, S. I., & Coleman, T. P. (2021). Data-driven noise modeling of digital DNA melting analysis enables prediction of sequence discriminating power.
Bioinformatics. https://doi.org/10.1093/bioinformatics/btaa1053
Kang, Q., Parkin, B., Giraldez, M. D., & Tewari, M. (2016). Mutant DNA quantification by digital PCR can be confounded by heating during DNA fragmentation.
BioTechniques. https://doi.org/10.2144/000114401
Swati, A., Mathuria, F., Bhavani, S., Malathy, E., & Mahadevan, R. (2017). A review on various encoding schemes used in digital DNA data storage. In
International Journal of Civil Engineering and Technology.
Cresci, S., Di Pietro, R., Petrocchi, M., Spognardi, A., & Tesconi, M. (2020). Emergent properties, models, and laws of behavioral similarities within groups of twitter users.
Computer Communications. https://doi.org/10.1016/j.comcom.2019.10.019
Becherer, L., Hess, J. F., Frischmann, S., Bakheit, M., Nitschko, H., Stinco, S., Zitz, F., Hofer, H., Porro, G., Hausladen, F., Stock, K., Drossart, D., Wurm, H., Kuhn, H., Huber, D., Hutzenlaub, T., Paust, N., Keller, M., Strohmeier, O., … Von Stetten, F. (2021). Point-of-care system for htlv-1 proviral load quantification by digital mediator displacement lamp.
Micromachines. https://doi.org/10.3390/mi12020159
Sombolestani, A. S., Cleenwerck, I., Cnockaert, M., Borremans, W., Wieme, A. D., De Vuyst, L., & Vandamme, P. (2021). Characterization of novel gluconobacter species from fruits and fermented food products: Gluconobacter cadivus sp. nov., gluconobacter vitians sp. nov. and gluconobacter potus sp. nov.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004751
Ma, Y. D., Chang, W. H., Luo, K., Wang, C. H., Liu, S. Y., Yen, W. H., & Lee, G. Bin. (2018). Digital quantification of DNA via isothermal amplification on a self-driven microfluidic chip featuring hydrophilic film-coated polydimethylsiloxane.
Biosensors and Bioelectronics. https://doi.org/10.1016/j.bios.2017.08.026
Choi, Y., Bae, H. J., Song, S., Kwon, S., & Park, W. (2016). DNA microdisk: Packaging and indexing of digital information stored DNA on encoded microparticle.
20th International Conference on Miniaturized Systems for Chemistry and Life Sciences, MicroTAS 2016.
Oguamanam, C., & Jain, V. (2017). Access and Benefit Sharing, Canadian and Aboriginal Research Ethics Policy After the Nagoya Protocol: Digital DNA and Transformations in Biotechnology.
Journal of Environmental Law and Practice.
Pavšič, J., Devonshire, A., Blejec, A., Foy, C. A., Van Heuverswyn, F., Jones, G. M., Schimmel, H., Žel, J., Huggett, J. F., Redshaw, N., Karczmarczyk, M., Mozioğlu, E., Akyürek, S., Akgöz, M., & Milavec, M. (2017). Inter-laboratory assessment of different digital PCR platforms for quantification of human cytomegalovirus DNA.
Analytical and Bioanalytical Chemistry. https://doi.org/10.1007/s00216-017-0206-0
Newman, A. M., Lovejoy, A. F., Klass, D. M., Kurtz, D. M., Chabon, J. J., Scherer, F., Stehr, H., Liu, C. L., Bratman, S. V., Say, C., Zhou, L., Carter, J. N., West, R. B., Sledge, G. W., Shrager, J. B., Loo, B. W., Neal, J. W., Wakelee, H. A., Diehn, M., & Alizadeh, A. A. (2016). Integrated digital error suppression for improved detection of circulating tumor DNA.
Nature Biotechnology. https://doi.org/10.1038/nbt.3520
Feng, G. Da, Chen, M. B., Zhang, X. J., Wang, D. D., & Zhu, H. H. (2019). Whole genome sequences reveal the presence of 11 heterotypic synonyms in the genus sphingobium and emended descriptions of sphingobium indicum, sphingobium fuliginis, sphingobium xenophagum and sphingobium cupriresistens.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.003432
Suehiro, Y., Zhang, Y., Hashimoto, S., Takami, T., Higaki, S., Shindo, Y., Suzuki, N., Hazama, S., Oka, M., Nagano, H., Sakaida, I., & Yamasaki, T. (2018). Highly sensitive faecal DNA testing of TWIST1 methylation in combination with faecal immunochemical test for haemoglobin is a promising marker for detection of colorectal neoplasia.
Annals of Clinical Biochemistry. https://doi.org/10.1177/0004563217691064
Cortes-Albayay, C., Dorador, C., Schumann, P., Schniete, J. K., Herron, P., Andrews, B., Asenjo, J., & Nouioui, I. (2019). Streptomyces altiplanensis sp. Nov., an alkalitolerant species isolated from chilean altiplano soil, and emended description of streptomyces chryseus (krasil’nikov et al. 1965) pridham 1970.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.003525
Pavšič, J., Žel, J., & Milavec, M. (2016). Digital PCR for direct quantification of viruses without DNA extraction.
Analytical and Bioanalytical Chemistry. https://doi.org/10.1007/s00216-015-9109-0
Huang, C.-H., Liou, J.-S., Wang, C.-L., & Huang, L. (2019). Draft Genome Sequence of Mediterraneibacter sp. nov. Strain gm002, Isolated from Human Feces .
Microbiology Resource Announcements. https://doi.org/10.1128/mra.00217-19
Wu, Z., Bai, Y., Cheng, Z., Liu, F., Wang, P., Yang, D., Li, G., Jin, Q., Mao, H., & Zhao, J. (2017). Absolute quantification of DNA methylation using microfluidic chip-based digital PCR.
Biosensors and Bioelectronics. https://doi.org/10.1016/j.bios.2017.05.021
Anderson, E. M., & Maldarelli, F. (2018). Quantification of HIV DNA Using Droplet Digital PCR Techniques.
Current Protocols in Microbiology. https://doi.org/10.1002/cpmc.62
Ruan, Q., Zou, F., Wang, Y., Zhang, Y., Xu, X., Lin, X., Tian, T., Zhang, H., Zhou, L., Zhu, Z., & Yang, C. (2021). Sensitive, rapid, and automated detection of DNA methylation based on digital microfluidics.
ACS Applied Materials and Interfaces. https://doi.org/10.1021/acsami.0c21995
Choi, G. M., Kim, K. M., Yun, C. S., Lee, S. Y., Kim, S. Y., Wee, J. H., & Im, W. T. (2020). Ochrobactrum soli sp. nov., Isolated from a Korean Cattle Farm.
Current Microbiology. https://doi.org/10.1007/s00284-020-01882-y
Hesselberth, J. R., Chen, X., Zhang, Z., Sabo, P. J., Sandstrom, R., Reynolds, A. P., Thurman, R. E., Neph, S., Kuehn, M. S., Noble, W. S., Fields, S., & Stamatoyannopoulos, J. A. (2009). Global mapping of protein-DNA interactions in vivo by digital genomic footprinting.
Nature Methods. https://doi.org/10.1038/nmeth.1313
Tohya, M., Watanabe, S., Tada, T., Tin, H. H., & Kirikae, T. (2020). Genome analysis-based reclassification of pseudomonas fuscovaginae and pseudomonas shirazica as later heterotypic synonyms of pseudomonas asplenii and pseudomonas asiatica, respectively.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004199
Travers, A. A., Muskhelishvili, G., & Thompson, J. M. T. (2012). DNA information: From digital code to analogue structure. In
Philosophical Transactions of the Royal Society A: Mathematical, Physical and Engineering Sciences. https://doi.org/10.1098/rsta.2011.0231
Pomari, E., Silva, R., Moro, L., Marca, G. La, Perandin, F., Verra, F., Bisoffi, Z., & Piubelli, C. (2020). Droplet digital PCR for the detection of plasmodium falciparum DNA in whole blood and serum: A comparative analysis with other molecular methods.
Pathogens. https://doi.org/10.3390/pathogens9060478
Liang, W., Xu, L., Sui, Z., Li, Y., Li, L., Wen, Y., Li, C., Ren, S., & Liu, G. (2016). Quantification of plasmid DNA reference materials for shiga toxin-producing escherichia coli based on UV, HR-ICP-MS and digital PCR.
Chemistry Central Journal. https://doi.org/10.1186/s13065-016-0201-0
Jeffreys, A. J., MacLeod, A., Tamaki, K., Neil, D. L., & Monckton, D. G. (1991). Minisatellite repeat coding as a digital approach to DNA typing.
Nature. https://doi.org/10.1038/354204a0
Fan, B., Blom, J., Klenk, H. P., & Borriss, R. (2017). Bacillus amyloliquefaciens, Bacillus velezensis, and Bacillus siamensis Form an “Operational Group B. amyloliquefaciens” within the B. subtilis species complex.
Frontiers in Microbiology. https://doi.org/10.3389/fmicb.2017.00022
von Ammon, U., Wood, S. A., Laroche, O., Zaiko, A., Lavery, S. D., Inglis, G. J., & Pochon, X. (2019). Linking Environmental DNA and RNA for Improved Detection of the Marine Invasive Fanworm Sabella spallanzanii.
Frontiers in Marine Science. https://doi.org/10.3389/fmars.2019.00621
Jackson, J. B., Choi, D. S., Luketich, J. D., Pennathur, A., Ståhlberg, A., & Godfrey, T. E. (2016). Multiplex Preamplification of Serum DNA to Facilitate Reliable Detection of Extremely Rare Cancer Mutations in Circulating DNA by Digital PCR.
Journal of Molecular Diagnostics. https://doi.org/10.1016/j.jmoldx.2015.10.004
Dong, L., Meng, Y., Sui, Z., Wang, J., Wu, L., & Fu, B. (2015). Comparison of four digital PCR platforms for accurate quantification of DNA copy number of a certified plasmid DNA reference material.
Scientific Reports. https://doi.org/10.1038/srep13174
Tanizawa, Y., Kobayashi, H., Kaminuma, E., Sakamoto, M., Ohkuma, M., Nakamura, Y., Arita, M., & Tohno, M. (2017). Genomic characterization reconfirms the taxonomic status of Lactobacillus parakefiri. In
Bioscience of Microbiota, Food and Health. https://doi.org/10.12938/bmfh.16-026
Nacheva, E., Mokretar, K., Soenmez, A., Pittman, A. M., Grace, C., Valli, R., Ejaz, A., Vattathil, S., Maserati, E., Houlden, H., Taanman, J. W., Schapira, A. H., & Proukakis, C. (2017). DNA isolation protocol effects on nuclear DNA analysis by microarrays, droplet digital PCR, and whole genome sequencing, and on mitochondrial DNA copy number estimation.
PLoS ONE. https://doi.org/10.1371/journal.pone.0180467
Thorell, K., Meier-Kolthoff, J. P., Sjöling, Å., & Martín-Rodríguez, A. J. (2019). Whole-genome sequencing redefines shewanella taxonomy.
Frontiers in Microbiology. https://doi.org/10.3389/fmicb.2019.01861
Gou, T., Hu, J., Wu, W., Ding, X., Zhou, S., Fang, W., & Mu, Y. (2018). Smartphone-based mobile digital PCR device for DNA quantitative analysis with high accuracy.
Biosensors and Bioelectronics. https://doi.org/10.1016/j.bios.2018.08.030
Wang, J., Yang, L., Diao, Y., Liu, J., Li, J., Li, R., Zheng, L., Zhang, K., Ma, Y., & Hao, X. (2021). Circulating tumour DNA methylation in hepatocellular carcinoma diagnosis using digital droplet PCR.
Journal of International Medical Research. https://doi.org/10.1177/0300060521992962
López, S. O., García-Olmo, D. C., García-Arranz, M., Guadalajara, H., Pastor, C., & García-Olmo, D. (2016). KRAS G12V mutation detection by droplet digital PCR in circulating cell-free DNA of colorectal cancer patients.
International Journal of Molecular Sciences. https://doi.org/10.3390/ijms17040484
Tardieu, H., Daly, D., Esteban-Lauzán, J., Hall, J., & Miller, G. (2020). Deliberately Digital Rewriting Enterprise DNA for Enduring Success. In
Springer.
Zhou, S., Gou, T., Hu, J., Wu, W., Ding, X., Fang, W., Hu, Z., & Mu, Y. (2019). A highly integrated real-time digital PCR device for accurate DNA quantitative analysis.
Biosensors and Bioelectronics. https://doi.org/10.1016/j.bios.2018.12.055
Aigrain, L., Gu, Y., & Quail, M. A. (2016). Quantitation of next generation sequencing library preparation protocol efficiencies using droplet digital PCR assays - a systematic comparison of DNA library preparation kits for Illumina sequencing.
BMC Genomics. https://doi.org/10.1186/s12864-016-2757-4
Bhat, S., & Emslie, K. R. (2016). Digital polymerase chain reaction for characterisation of DNA reference materials. In
Biomolecular Detection and Quantification. https://doi.org/10.1016/j.bdq.2016.04.001
Li, X., Tambong, J., Yuan, K. X., Chen, W., Xu, H., André Lévesque, C., & De Boer, S. H. (2018). Re-classification of clavibacter michiganensis subspecies on the basis of whole-genome and multi-locus sequence analyses.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.002492
van Ginkel, J. H., van den Broek, D. A., van Kuik, J., Linders, D., de Weger, R., Willems, S. M., & Huibers, M. M. H. (2017). Preanalytical blood sample workup for cell-free DNA analysis using Droplet Digital PCR for future molecular cancer diagnostics.
Cancer Medicine. https://doi.org/10.1002/cam4.1184
Tsaftaris, S. A., & Katsaggelos, A. K. (2009). Retrieval efficiency of DNA-based databases of digital signals.
IEEE Transactions on Nanobioscience. https://doi.org/10.1109/TNB.2009.2026371
Suzuki, M., Umeda, K., Kimura, M., Imaoka, K., Morikawa, S., & Maeda, K. (2020). Capnocytophaga felis sp. Nov. isolated from the feline oral cavity.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004176
Demuth, C., Spindler, K. L. G., Johansen, J. S., Pallisgaard, N., Nielsen, D., Hogdall, E., Vittrup, B., & Sorensen, B. S. (2018). Measuring KRAS Mutations in Circulating Tumor DNA by Droplet Digital PCR and Next-Generation Sequencing.
Translational Oncology. https://doi.org/10.1016/j.tranon.2018.07.013
Lun, F. M. F., Chiu, R. W. K., Chan, K. C. A., Tak, Y. L., Tze, K. L., & Lo, Y. M. D. (2008). Microfluidics digital PCR reveals a higher than expected fraction of fetal DNA in maternal plasma.
Clinical Chemistry. https://doi.org/10.1373/clinchem.2008.111385
Lin, S. Y., Tsai, C. F., Hameed, A., & Young, C. C. (2020). Cerasibacillus terrae sp. nov., isolated from maize field, and emended description of Cerasibacillus quisquiliarum Nakamura et al. 2004.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004470
Brychta, N., Krahn, T., & Von Ahsen, O. (2016). Detection of KRAS mutations in circulating tumor DNA by digital PCR in early stages of pancreatic cancer.
Clinical Chemistry. https://doi.org/10.1373/clinchem.2016.257469
Athitha M A Akshatha M A, V. B. (2014). A Review on DNA Based Cryptographic Techniques.
International Journal of Science and Research (IJSR).
Zhou, H., & Huan, X. (2019). Applying CRISPR-Cas9 off-target editing on DNA based steganography.
International Journal of Advanced Computer Science and Applications. https://doi.org/10.14569/ijacsa.2019.0100801
Tian, Q., Yu, B., Mu, Y., Xu, Y., Ma, C., Zhang, T., Jin, W., & Jin, Q. (2015). An integrated temporary negative pressure assisted microfluidic chip for DNA isolation and digital PCR detection.
RSC Advances. https://doi.org/10.1039/c5ra18166f
van Ginkel, J. H., Huibers, M. M. H., van Es, R. J. J., de Bree, R., & Willems, S. M. (2017). Droplet digital PCR for detection and quantification of circulating tumor DNA in plasma of head and neck cancer patients.
BMC Cancer. https://doi.org/10.1186/s12885-017-3424-0
Tripodi, L., Witters, D., Kokalj, T., Huber, H. J., Puers, R., Lammertyn, J., & Spasic, D. (2018). Sub-femtomolar detection of DNA and discrimination of mutant strands using microwell-array assisted digital enzyme-linked oligonucleotide assay.
Analytica Chimica Acta. https://doi.org/10.1016/j.aca.2018.08.056
Corbisier, P., Pinheiro, L., Mazoua, S., Kortekaas, A. M., Chung, P. Y. J., Gerganova, T., Roebben, G., Emons, H., & Emslie, K. (2015). DNA copy number concentration measured by digital and droplet digital quantitative PCR using certified reference materials.
Analytical and Bioanalytical Chemistry. https://doi.org/10.1007/s00216-015-8458-z
Dimopoulou, M., Antonini, M., Barbry, P., & Appuswamy, R. (2020). Storing digital data into DNA: A comparative study of quaternary code construction.
ICASSP, IEEE International Conference on Acoustics, Speech and Signal Processing - Proceedings. https://doi.org/10.1109/ICASSP40776.2020.9054654
Li, S. H., Song, J., Lim, Y., Joung, Y., Kang, I., & Cho, J. C. (2020). Halioglobus maricola sp. nov., isolated from coastal seawater.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.003985
Termanini, R. (2020). The digital universe with DNA—the magic of CRISPR. In
Storing Digital Binary Data in Cellular DNA. https://doi.org/10.1016/b978-0-12-823295-8.00005-7
Martinelli, E. M., Farioli, M. C., & Tunisini, A. (2020). New companies’ DNA: the heritage of the past industrial revolutions in digital transformation.
Journal of Management and Governance. https://doi.org/10.1007/s10997-020-09539-5
Prachumwat, A., Wechprasit, P., Srisala, J., Kriangsaksri, R., Flegel, T. W., Thitamadee, S., & Sritunyalucksana, K. (2020). Shewanella khirikhana sp. nov. – a shrimp pathogen isolated from a cultivation pond exhibiting early mortality syndrome.
Microbial Biotechnology. https://doi.org/10.1111/1751-7915.13538
Cresci, S., Di Pietro, R., Petrocchi, M., Spognardi, A., & Tesconi, M. (2016). DNA-Inspired Online Behavioral Modeling and Its Application to Spambot Detection.
IEEE Intelligent Systems. https://doi.org/10.1109/MIS.2016.29
Yamamoto, M., Ushio, R., Watanabe, H., Tachibana, T., Tanaka, M., Yokose, T., Tsukiji, J., Nakajima, H., & Kaneko, T. (2018). Detection of Mycobacterium tuberculosis-derived DNA in circulating cell-free DNA from a patient with disseminated infection using digital PCR.
International Journal of Infectious Diseases. https://doi.org/10.1016/j.ijid.2017.11.018
Liu, A., Zhang, Y. J., Cheng, P., Peng, Y. J., Blom, J., & Xue, Q. J. (2020). Whole genome analysis calls for a taxonomic rearrangement of the genus Colwellia.
Antonie van Leeuwenhoek, International Journal of General and Molecular Microbiology. https://doi.org/10.1007/s10482-020-01405-6
Li, N., Dhilipkannah, P., & Jiang, F. (2021). High-throughput detection of multiple miRNAs and methylated DNA by droplet digital PCR.
Journal of Personalized Medicine. https://doi.org/10.3390/jpm11050359
Gutteridge, A., Rathbone, V. M., Gibbons, R., Bi, M., Archard, N., Davies, K. E. J., Brown, J., Plagnol, V., Pillay, N., Amary, F., O’Donnell, P., Gupta, M., Tirabosco, R., Flanagan, A. M., & Forshew, T. (2017). Digital PCR analysis of circulating tumor DNA: a biomarker for chondrosarcoma diagnosis, prognostication, and residual disease detection.
Cancer Medicine. https://doi.org/10.1002/cam4.1146
Ramos, P., Handt, O., & Taylor, D. (2020). Investigating the position and level of DNA transfer to undergarments during digital sexual assault.
Forensic Science International: Genetics. https://doi.org/10.1016/j.fsigen.2020.102316
Cao, Z., Wu, W., Wei, H., Gao, C., Zhang, L., Wu, C., & Hou, L. (2020). Using droplet digital PCR in the detection of Mycobacterium tuberculosis DNA in FFPE samples.
International Journal of Infectious Diseases. https://doi.org/10.1016/j.ijid.2020.07.045
Pharo, H. D., Andresen, K., Berg, K. C. G., Lothe, R. A., Jeanmougin, M., & Lind, G. E. (2018). A robust internal control for high-precision DNA methylation analyses by droplet digital PCR.
Clinical Epigenetics. https://doi.org/10.1186/s13148-018-0456-5
Didelot, A., Kotsopoulos, S. K., Lupo, A., Pekin, D., Li, X., Atochin, I., Srinivasan, P., Zhong, Q., Olson, J., Link, D. R., Laurent-Puig, P., Blons, H., Hutchison, J. B., & Taly, V. (2013). Multiplex picoliter-droplet digital PCR for quantitative assessment of DNA integrity in clinical samples.
Clinical Chemistry. https://doi.org/10.1373/clinchem.2012.193409
Whale, A. S., Cowen, S., Foy, C. A., & Huggett, J. F. (2013). Methods for Applying Accurate Digital PCR Analysis on Low Copy DNA Samples.
PLoS ONE. https://doi.org/10.1371/journal.pone.0058177
Bruni, I., De Mattia, F., Martellos, S., Galimberti, A., Savadori, P., Casiraghi, M., Nimis, P. L., & Labra, M. (2012). DNA barcoding as an effective tool in improving a digital plant identification system: A case study for the area of Mt. Valerio, Trieste (NE Italy).
PLoS ONE. https://doi.org/10.1371/journal.pone.0043256
Wood-Bouwens, C. M., Haslem, D., Moulton, B., Almeda, A. F., Lee, H., Heestand, G. M., Nadauld, L. D., & Ji, H. P. (2020). Therapeutic Monitoring of Circulating DNA Mutations in Metastatic Cancer with Personalized Digital PCR.
Journal of Molecular Diagnostics. https://doi.org/10.1016/j.jmoldx.2019.10.008
Herbst, A., Widjaja, K., Nguy, B., Lushaj, E. B., Moore, T. M., Hevener, A. L., McKenzie, D., Aiken, J. M., & Wanagat, J. (2017). Digital PCR Quantitation of Muscle Mitochondrial DNA: Age, Fiber Type, and Mutation-Induced Changes.
Journals of Gerontology - Series A Biological Sciences and Medical Sciences. https://doi.org/10.1093/gerona/glx058
Carpentieri, B. (2020). Compression of next-generation sequencing data and of DNA digital files.
Algorithms. https://doi.org/10.3390/A13060151
Alikian, M., Ellery, P., Forbes, M., Gerrard, G., Kasperaviciute, D., Sosinsky, A., Mueller, M., Whale, A. S., Milojkovic, D., Apperley, J., Huggett, J. F., Foroni, L., & Reid, A. G. (2016). Next-Generation Sequencing-Assisted DNA-Based Digital PCR for a Personalized Approach to the Detection and Quantification of Residual Disease in Chronic Myeloid Leukemia Patients.
Journal of Molecular Diagnostics. https://doi.org/10.1016/j.jmoldx.2015.09.005
Alikian, M., Ellery, P., Forbes, M., Gerrard, G., Kasperaviciute, D., Sosinsky, A., Mueller, M., Whale, A. S., Milojkovic, D., Apperley, J., Huggett, J. F., Foroni, L., & Reid, A. G. (2016). Next-Generation Sequencing-Assisted DNA-Based Digital PCR for a Personalized Approach to the Detection and Quantification of Residual Disease in Chronic Myeloid Leukemia Patients.
Journal of Molecular Diagnostics. https://doi.org/10.1016/j.jmoldx.2015.09.005
Daems, D., Rutten, I., Bath, J., Decrop, D., Van Gorp, H., Ruiz, E. P., De Feyter, S., Turberfield, A. J., & Lammertyn, J. (2019). Controlling the bioreceptor spatial distribution at the nanoscale for single molecule counting in microwell arrays.
ACS Sensors. https://doi.org/10.1021/acssensors.9b00877
Ye, W., Tang, X., Liu, C., Wen, C., Li, W., & Lyu, J. (2017). Accurate quantitation of circulating cell-free mitochondrial DNA in plasma by droplet digital PCR.
Analytical and Bioanalytical Chemistry. https://doi.org/10.1007/s00216-017-0217-x
Flanagan, N., & McAlister, C. (2011). The transfer and persistence of DNA under the fingernails following digital penetration of the vagina.
Forensic Science International: Genetics. https://doi.org/10.1016/j.fsigen.2010.10.008
Sun, L., He, J., Luo, J., & Coy, D. H. (2019). iMedPub Journals DNA and the Digital Data Storage.
Health Science Journal.
Hardinge, P., Baxani, D. K., McCloy, T., Murray, J. A. H., & Castell, O. K. (2020). Bioluminescent detection of isothermal DNA amplification in microfluidic generated droplets and artificial cells.
Scientific Reports. https://doi.org/10.1038/s41598-020-78996-7
Geng, H., Zhou, C., & Guo, C. (2019). DNA-based digital comparator systems constructed by multifunctional nanoswitches.
Nanoscale. https://doi.org/10.1039/c9nr08216f
Macgregor-Das, A., Yu, J., Tamura, K., Abe, T., Suenaga, M., Shindo, K., Borges, M., Koi, C., Kohi, S., Sadakari, Y., Dal Molin, M., Almario, J. A., Ford, M., Chuidian, M., Burkhart, R., He, J., Hruban, R. H., Eshleman, J. R., Klein, A. P., … Goggins, M. (2020). Detection of Circulating Tumor DNA in Patients with Pancreatic Cancer Using Digital Next-Generation Sequencing.
Journal of Molecular Diagnostics. https://doi.org/10.1016/j.jmoldx.2020.02.010
Zhang, H., Sanin, C., & Szczerbicki, E. (2012). The development of decisional DNA DIGITAL TV.
Frontiers in Artificial Intelligence and Applications. https://doi.org/10.3233/978-1-61499-105-2-1500
Takano, S., Fukasawa, M., Shindo, H., Takahashi, E., Fukasawa, Y., Kawakami, S., Hayakawa, H., Kuratomi, N., Kadokura, M., Maekawa, S., & Enomoto, N. (2021). Digital next-generation sequencing of cell-free DNA for pancreatic cancer.
JGH Open. https://doi.org/10.1002/jgh3.12530
Lee, H. H., Kalhor, R., Goela, N., Bolot, J., & Church, G. M. (2019). Supplementary Materials for Terminator-free template-independent enzymatic DNA synthesis for digital information storage Table of Contents.
Nature Communications.
Laurito, M., Ontivero, I. M., & Almirón, W. R. (2019). Increasing the digital repository of DNA barcoding sequences of sand flies (Psychodidae: Phlebotominae).
Memorias Do Instituto Oswaldo Cruz. https://doi.org/10.1590/0074-02760190208
Tomida, J., Akiyama-Miyoshi, T., Tanaka, K., Hayashi, M., Kutsuna, R., Fujiwara, N., & Kawamura, Y. (2021). Fusobacterium watanabei sp. nov. As additional species within the genus Fusobacerium, isolated from human clinical specimens.
Anaerobe. https://doi.org/10.1016/j.anaerobe.2021.102323
Burjanivova, T., Malicherova, B., Grendar, M., Minarikova, E., Dusenka, R., Vanova, B., Bobrovska, M., Pecova, T., Homola, I., Lasabova, Z., & Plank, L. (2019). Detection of BRAFV600E Mutation in Melanoma Patients by Digital PCR of Circulating DNA.
Genetic Testing and Molecular Biomarkers. https://doi.org/10.1089/gtmb.2018.0193
Li, H., Bai, R., Zhao, Z., Tao, L., Ma, M., Ji, Z., Jian, M., Ding, Z., Dai, X., Bao, F., & Liu, A. (2018). Application of droplet digital PCR to detect the pathogens of infectious diseases. In
Bioscience Reports. https://doi.org/10.1042/BSR20181170
Camara, A., Konate, S., Lo, C. I., Kuete, E., Sarr, M., Amsrtong, N., Niare, S., Thera, M. A., Fenollar, F., Raoult, D., & Million, M. (2020). Virgibacillus ihumii sp. nov., a new bacterium isolated from the stool of healthy African children.
New Microbes and New Infections. https://doi.org/10.1016/j.nmni.2020.100790
Yukphan, P., Charoenyingcharoen, P., Malimas, S., Muramatsu, Y., Nakagawa, Y., Tanasupawat, S., & Yamada, Y. (2020). Gluconobacter aidae sp. Nov., an acetic acid bacteria isolated from tropical fruits in Thailand.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004292
Zhang, X., Zhang, L., Yu, X., Zhang, J., Jiao, Y., Ju, H., Wang, X., Zhao, J., & Xiang, W. (2021). Nocardia bovistercoris sp. Nov., an actinobacterium isolated from cow dung.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004699
Agarwal, N., Mahendran, A., & Lakshmanan, R. (2019). Trusted third party auditing for cloud security using digital signature and dna cryptography.
International Journal of Scientific and Technology Research.
Yim, S. S., McBee, R. M., Song, A. M., Huang, Y., Sheth, R. U., & Wang, H. H. (2021). Robust direct digital-to-biological data storage in living cells.
Nature Chemical Biology. https://doi.org/10.1038/s41589-020-00711-4
Freedman, G. (2014). Google
TM versus me
TM: Who owns the rights to my digital DNA?
Policy Futures in Education. https://doi.org/10.2304/pfie.2014.12.4.482
Madhaiyan, M., Saravanan, V. S., Wirth, J. S., & Whitman, W. B. (2020). Reclassification of sphingomonas aeria as a later heterotypic synonym of sphingomonas carotinifaciens based on whole-genome sequence analysis.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004045
Tian, Q., Mu, Y., Xu, Y., Song, Q., Yu, B., Ma, C., Jin, W., & Jin, Q. (2015). An integrated microfluidic system for bovine DNA purification and digital PCR detection.
Analytical Biochemistry. https://doi.org/10.1016/j.ab.2015.08.030
Li, G., Zhu, L., Wu, Z., He, Y., Tan, H., & Sun, S. (2016). Digital concentration readout of DNA by absolute quantification of optically countable gold nanorods.
Analytical Chemistry. https://doi.org/10.1021/acs.analchem.6b02712
Sidstedt, M., Rådström, P., & Hedman, J. (2020). PCR inhibition in qPCR, dPCR and MPS—mechanisms and solutions. In
Analytical and Bioanalytical Chemistry. https://doi.org/10.1007/s00216-020-02490-2
Zhang, Y., Xu, Y., Zhong, W., Zhao, J., Chen, M., Zhang, L., Li, L., & Wang, M. (2017). Total DNA input is a crucial determinant of the sensitivity of plasma cell-free DNA EGFR mutation detection using droplet digital PCR.
Oncotarget. https://doi.org/10.18632/oncotarget.14390
Li, G., Zhu, L., He, Y., Tan, H., & Sun, S. (2017). Digital triplex DNA assay based on plasmonic nanocrystals.
Analytical and Bioanalytical Chemistry. https://doi.org/10.1007/s00216-017-0307-9
Tchagang, C. F., Xu, R., Doumbou, C. L., & Tambong, J. T. (2018). Genome analysis of two novel Pseudomonas strains exhibiting differential hypersensitivity reactions on tobacco seedlings reveals differences in nonflagellar T3SS organization and predicted effector proteins.
MicrobiologyOpen. https://doi.org/10.1002/mbo3.553
Montagnier, L., Del Giudice, E., Aïssa, J., Lavallee, C., Motschwiller, S., Capolupo, A., Polcari, A., Romano, P., Tedeschi, A., & Vitiello, G. (2015). Transduction of DNA information through water and electromagnetic waves.
Electromagnetic Biology and Medicine. https://doi.org/10.3109/15368378.2015.1036072
Perreten, V., Kania, S. A., & Bemis, D. (2020). Staphylococcus ursi sp. Nov., a new member of the ‘staphylococcus intermedius group’ isolated from healthy black bears.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004324
Dube, S., Qin, J., & Ramakrishnan, R. (2008). Mathematical analysis of copy number variation in a DNA sample using digital PCR on a nanofluidic device.
PLoS ONE. https://doi.org/10.1371/journal.pone.0002876
Komaki, H., & Tamura, T. (2020). Reclassification of streptomyces hygroscopicus subsp. Glebosus and streptomyces libani subsp. rufus as later heterotypic synonyms of streptomyces platensis.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004279
Schoepp, N. G., Khorosheva, E. M., Schlappi, T. S., Curtis, M. S., Humphries, R. M., Hindler, J. A., & Ismagilov, R. F. (2016). Digital Quantification of DNA Replication and Chromosome Segregation Enables Determination of Antimicrobial Susceptibility after only 15 Minutes of Antibiotic Exposure.
Angewandte Chemie - International Edition. https://doi.org/10.1002/anie.201602763
Fernando, M. R., Jiang, C., Krzyzanowski, G. D., & Ryan, W. L. (2018). Analysis of human blood plasma cell-free DNA fragment size distribution using EvaGreen chemistry based droplet digital PCR assays.
Clinica Chimica Acta. https://doi.org/10.1016/j.cca.2018.04.017
Athamanolap, P., Hsieh, K., O’Keefe, C. M., Zhang, Y., Yang, S., & Wang, T. H. (2019). Nanoarray Digital Polymerase Chain Reaction with High-Resolution Melt for Enabling Broad Bacteria Identification and Pheno-Molecular Antimicrobial Susceptibility Test.
Analytical Chemistry. https://doi.org/10.1021/acs.analchem.9b02344
Zhang, Y., Ge, C., Zhu, C., & Salaita, K. (2014). DNA-based digital tension probes reveal integrin forces during early cell adhesion.
Nature Communications. https://doi.org/10.1038/ncomms6167
Duewer, D. L., Kline, M. C., Romsos, E. L., & Toman, B. (2018). Evaluating droplet digital PCR for the quantification of human genomic DNA: converting copies per nanoliter to nanograms nuclear DNA per microliter.
Analytical and Bioanalytical Chemistry. https://doi.org/10.1007/s00216-018-0982-1
Phạm, H. T. T., Suwannapan, W., Koomsiri, W., Inahashi, Y., Také, A., Matsumoto, A., & Thamchaipenet, A. (2020). Fodinicola acaciae sp. nov., an endophytic actinomycete isolated from the roots of Acacia mangium willd. and its genome analysis.
Microorganisms. https://doi.org/10.3390/microorganisms8040467
Zur, O., & Walker, A. (2011). On Digital Immigrants and Digital Natives : How the Digital Divide Affects Families , Educational Institutions , and the Workplace.
Zurinstitute.
Trinh, N. H., & Kim, J. (2020). Paraburkholderia flava sp. Nov., isolated from cool temperatforest soil.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004063
Cresci, S., Di Pietro, R., Petrocchi, M., Spognardi, A., & Tesconi, M. (2017). Exploiting digital DNA for the analysis of similarities in twitter behaviours.
Proceedings - 2017 International Conference on Data Science and Advanced Analytics, DSAA 2017. https://doi.org/10.1109/DSAA.2017.57
Peitz, C., Sprüssel, A., Linke, R. B., Astrahantseff, K., Grimaldi, M., Schmelz, K., Toedling, J., Schulte, J. H., Fischer, M., Messerschmidt, C., Beule, D., Keilholz, U., Eggert, A., Deubzer, H. E., & Lodrini, M. (2020). Multiplexed Quantification of Four Neuroblastoma DNA Targets in a Single Droplet Digital PCR Reaction.
Journal of Molecular Diagnostics. https://doi.org/10.1016/j.jmoldx.2020.07.006
Madhaiyan, M., See-Too, W. S., Ee, R., Saravanan, V. S., Wirth, J. S., Alex, T. H. H., Lin, C., Kim, S. J., Weon, H. Y., Kwon, S. W., Whitman, W. B., & Ji, L. (2020). Chitinasiproducens palmae gen. Nov., sp. nov., a new member of the family burkholderiaceae isolated from leaf tissues of oil palm (elaeis guineensis jacq.).
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004084
Sidstedt, M., Hedman, J., Romsos, E. L., Waitara, L., Wadsö, L., Steffen, C. R., Vallone, P. M., & Rådström, P. (2018). Inhibition mechanisms of hemoglobin, immunoglobulin G, and whole blood in digital and real-time PCR.
Analytical and Bioanalytical Chemistry. https://doi.org/10.1007/s00216-018-0931-z
Huang, C. H., Li, S. W., Huang, L., & Watanabe, K. (2018). Identification and classification for the Lactobacillus casei group.
Frontiers in Microbiology. https://doi.org/10.3389/fmicb.2018.01974
Farzadfard, F., Gharaei, N., Higashikuni, Y., Jung, G., Cao, J., & Lu, T. K. (2019). Single-Nucleotide-Resolution Computing and Memory in Living Cells.
Molecular Cell. https://doi.org/10.1016/j.molcel.2019.07.011
Lou, Q., & Jiang, L. (2018). BRAWL: A Spintronics-Based Portable Basecalling-in-Memory Architecture for Nanopore Genome Sequencing.
IEEE Computer Architecture Letters. https://doi.org/10.1109/LCA.2018.2882384
Anani, H., Abou Abdallah, R., Chelkha, N., Fontanini, A., Ricaboni, D., Mailhe, M., Raoult, D., & Fournier, P. E. (2019). Draft genome and description of Merdibacter massiliensis gen.nov., sp. nov., a new bacterium genus isolated from the human ileum.
Scientific Reports. https://doi.org/10.1038/s41598-019-44343-8
Iwama, E., Sakai, K., Azuma, K., Harada, T., Harada, D., Nosaki, K., Hotta, K., Ohyanagi, F., Kurata, T., Fukuhara, T., Akamatsu, H., Goto, K., Shimose, T., Kishimoto, J., Nakanishi, Y., Nishio, K., & Okamoto, I. (2017). Monitoring of somatic mutations in circulating cell-free DNA by digital PCR and next-generation sequencing during afatinib treatment in patients with lung adenocarcinoma positive for EGFR activating mutations.
Annals of Oncology. https://doi.org/10.1093/annonc/mdw531
Oberhettinger, P., Schüle, L., Marschal, M., Bezdan, D., Ossowski, S., Dörfel, D., Vogel, W., Rossen, J. W., Willmann, M., & Peter, S. (2020). Description of citrobacter cronae sp. Nov., isolated from human rectal swabs and stool samples.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004100
Tambong, J. T., Xu, R., & Bromfield, E. S. P. (2017). Pseudomonas canadensis sp. Nov., a biological control agent isolated from a field plot under long-term mineral fertilization. In
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.001698
Dutra, L., Franz, O., Puupponen, V. M., & Tiirola, M. (2020). DNA recovery from Droplet Digital
TM PCR emulsions using liquid nitrogen.
BioTechniques. https://doi.org/10.2144/btn-2020-0076
Regan, J., & Karlin-Neumann, G. (2018). Phasing DNA markers using digital PCR. In
Methods in Molecular Biology. https://doi.org/10.1007/978-1-4939-7778-9_28
Nain, Z., Rana, H. K., Liò, P., Islam, S. M. S., Summers, M. A., & Moni, M. A. (2021). Pathogenetic profiling of COVID-19 and SARS-like viruses.
Briefings in Bioinformatics. https://doi.org/10.1093/bib/bbaa173
Kuncharoen, N., Kudo, T., Yuki, M., Okuma, M., Pittayakhajonwut, P., & Tanasupawat, S. (2019). Micromonospora radicis sp. Nov., isolated from roots of Azadirachta indica var. siamensis Valenton, and reclassification of Jishengella zingiberis as Micromonospora zingiberis comb. nov.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.003574
Mullins, A. J., Li, Y., Qin, L., Hu, X., Xie, L., Gu, C., Mahenthiralingam, E., Liao, X., & Webster, G. (2020). Reclassification of the biocontrol agents bacillus subtilis by-2 and tu-100 as bacillus velezensis and insights into the genomic and specialized metabolite diversity of the species.
Microbiology (United Kingdom). https://doi.org/10.1099/mic.0.000986
Hematpour, N., Ahadpour, S., & Behnia, S. (2021). Presence of dynamics of quantum dots in the digital signature using DNA alphabet and chaotic S-box.
Multimedia Tools and Applications. https://doi.org/10.1007/s11042-020-10059-5
Dobbs, F. M., Van Eijk, P., Fellows, M. D., Loiacono, L., Nitsch, R., & Reed, S. H. (2020). Precision digital mapping of endogenous and induced genomic DNA breaks by INDUCE-seq.
BioRxiv.
Cerezales, M., Xanthopoulou, K., Ertel, J., Nemec, A., Bustamante, Z., Seifert, H., Gallego, L., & Higgins, P. G. (2018). Identification of acinetobacter seifertii isolated from bolivian hospitals.
Journal of Medical Microbiology. https://doi.org/10.1099/jmm.0.000751
Sofronova, J. K., Ilinsky, Y. Y., Orishchenko, K. E., Chupakhin, E. G., Lunev, E. A., & Mazunin, I. O. (2016). Detection of mutations in mitochondrial DNA by droplet digital PCR.
Biochemistry (Moscow). https://doi.org/10.1134/S0006297916100011
Meltzer, R. H., Krogmeier, J. R., Kwok, L. W., Allen, R., Crane, B., Griffis, J. W., Knaian, L., Kojanian, N., Malkin, G., Nahas, M. K., Papkov, V., Shaikh, S., Vyavahare, K., Zhong, Q., Zhou, Y., Larson, J. W., & Gilmanshin, R. (2011). A lab-on-chip for biothreat detection using single-molecule DNA mapping.
Lab on a Chip. https://doi.org/10.1039/c0lc00477d
Kim, H., Bartsch, M. S., Renzi, R. F., He, J., Van de Vreugde, J. L., Claudnic, M. R., & Patel, K. D. (2011). Automated Digital Microfluidic Sample Preparation for Next-Generation DNA Sequencing.
Journal of Laboratory Automation. https://doi.org/10.1016/j.jala.2011.07.001
Intra, B., Panbangred, W., Inahashi, Y., Také, A., Mori, M., Ōmura, S., & Matsumoto, A. (2020). Micromonospora pelagivivens sp. nov., a new species of the genus micromonospora isolated from deep-sea sediment in Japan.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004136
Arantes, A. L., Moreira, J. P. C., Diender, M., Parshina, S. N., Stams, A. J. M., Alves, M. M., Alves, J. I., & Sousa, D. Z. (2020). Enrichment of Anaerobic Syngas-Converting Communities and Isolation of a Novel Carboxydotrophic Acetobacterium wieringae Strain JM.
Frontiers in Microbiology. https://doi.org/10.3389/fmicb.2020.00058
Lee, J., Kim, Y., Kim, C., & Ree, M. (2017). New high performance digital memory devices fabricated with DNA and DNA-mimics.
Materials Horizons. https://doi.org/10.1039/c7mh00028f
Hidese, R., Kawato, K., Nakura, Y., Fujiwara, A., Yasukawa, K., Yanagihara, I., & Fujiwara, S. (2018). Thermostable DNA helicase improves the sensitivity of digital PCR.
Biochemical and Biophysical Research Communications. https://doi.org/10.1016/j.bbrc.2017.12.053
Herbreteau, G., Vallée, A., Knol, A. C., Théoleyre, S., Quéreux, G., Varey, E., Khammari, A., Dréno, B., & Denis, M. G. (2018). Quantitative monitoring of circulating tumor DNA predicts response of cutaneous metastatic melanoma to anti-PD1 immunotherapy.
Oncotarget. https://doi.org/10.18632/oncotarget.25404
Baker, C. S., Steel, D., Nieukirk, S., & Klinck, H. (2018). Environmental DNA (eDNA) from the wake of the whales: Droplet digital PCR for detection and species identification.
Frontiers in Marine Science. https://doi.org/10.3389/fmars.2018.00133
Zhang, L., Zhou, X. Y., Su, X. J., Hu, Q., & Jiang, J. D. (2019). Spirosoma sordidisoli sp. nov., a propanil-degrading bacterium isolated from a herbicide-contaminated soil.
Antonie van Leeuwenhoek, International Journal of General and Molecular Microbiology. https://doi.org/10.1007/s10482-019-01278-4
Tohya, M., Watanabe, S., Teramoto, K., Tada, T., Kuwahara-Arai, K., Mya, S., Zin, K. N., Kirikae, T., & Tin, H. H. (2020). Pseudomonas yangonensis sp. nov., isolated from wound samples of patients in a hospital in Myanmar.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/IJSEM.0.004181
Momtaz, P., Pentsova, E., Abdel-Wahab, O., Diamond, E., Hyman, D., Merghoub, T., You, D., Gasmi, B., Viale, A., & Chapman, P. B. (2016). Quantification of tumor-derived cell free DNA(cfDNA) by digital PCR (DigPCR) in cerebrospinal fluid of patients with BRAFV600 mutated malignancies.
Oncotarget. https://doi.org/10.18632/oncotarget.13397
Sorber, L., Zwaenepoel, K., Deschoolmeester, V., Roeyen, G., Lardon, F., Rolfo, C., & Pauwels, P. (2017). A Comparison of Cell-Free DNA Isolation Kits: Isolation and Quantification of Cell-Free DNA in Plasma.
Journal of Molecular Diagnostics. https://doi.org/10.1016/j.jmoldx.2016.09.009
Chang-Hao Tsao, S., Weiss, J., Hudson, C., Christophi, C., Cebon, J., Behren, A., & Dobrovic, A. (2015). Monitoring response to therapy in melanoma by quantifying circulating tumour DNA with droplet digital PCR for BRAF and NRAS mutations.
Scientific Reports. https://doi.org/10.1038/srep11198
Jung, S. W., Min, H. K., Kim, Y. H., Choi, H. A., Lee, S. Y., Bae, Y. J., & Paek, W. K. (2016). A DNA barcode library of the beetle reference collection (Insecta: Coleoptera) in the National Science Museum, Korea.
Journal of Asia-Pacific Biodiversity. https://doi.org/10.1016/j.japb.2016.03.005
Oosthuizen, L., Charimba, G., Hitzeroth, A., Nde, A. L., Steyn, L., Newman, J., & Hugo, C. (2019). Chryseobacterium pennipullorum sp. Nov., isolated from poultry feather waste.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.003491
Gallegos, J. E., Kar, D. M., Ray, I., Ray, I., & Peccoud, J. (2020). Securing the Exchange of Synthetic Genetic Constructs Using Digital Signatures.
ACS Synthetic Biology. https://doi.org/10.1021/acssynbio.0c00401
Garofalo, S. G., & Farenga, S. J. (2021). Cognition and Spatial Concept Formation: Comparing Non-digital and Digital Instruction Using Three-Dimensional Models in Science.
Technology, Knowledge and Learning. https://doi.org/10.1007/s10758-019-09425-6
Tohya, M., Watanabe, S., Teramoto, K., Tada, T., Kuwahara-Arai, K., Mya, S., Zin, K. N., Kirikae, T., & Tin, H. H. (2020). Pseudomonas yangonensis sp. nov., isolated from wound samples of patients in a hospital in Myanmar.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/IJSEM.0.004181
Sun, J., Wang, Q., Diao, W., Zhou, C., Wang, B., Rao, L., & Yang, P. (2019). Digital information storage on DNA in living organisms. In
Medical Research Archives Jian Sun et al. Medical Research Archives.
Jiang, W. K., Gao, Q. Q., Zhang, L., Sun, G. J., Zhang, M. L., Liu, X. A., Wang, H., Zhou, Y. D., Ke, Z. J., & Hong, Q. (2020). Ornithinicoccus soli sp. Nov., isolated from farmland soil.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.003972
Sawada, H., Fujikawa, T., Nishiwaki, Y., & Horita, H. (2020). Pseudomonas kitaguniensis sp. Nov., a pathogen causing bacterial rot of welsh onion in japan.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004123
Kline, M. C., & Duewer, D. L. (2020). Evaluating digital PCR for the quantification of human nuclear DNA: determining target strandedness.
Analytical and Bioanalytical Chemistry. https://doi.org/10.1007/s00216-020-02733-2
An, H. T., Houchaimi, S., Burkhart, C. T., & Schertzer, M. J. (2020). Dna ligation on a digital microfluidic device.
ASME 2020 18th International Conference on Nanochannels, Microchannels, and Minichannels, ICNMM 2020, Collocated with the ASME 2020 Heat Transfer Summer Conference and the ASME 2020 Fluids Engineering Division Summer Meeting. https://doi.org/10.1115/ICNMM2020-1028
Li, M., Dong, C., Law, M. K., Jia, Y., Mak, P. I., & Martins, R. P. (2019). Hydrodynamic-flow-enhanced rapid mixer for isothermal DNA hybridization kinetics analysis on digital microfluidics platform.
Sensors and Actuators, B: Chemical. https://doi.org/10.1016/j.snb.2019.02.021
Tamura, T., Saito, S., Hamada, M., Kang, Y., Hoshino, Y., Gonoi, T., Mikami, Y., & Yaguchi, T. (2020). Gordonia crocea sp. Nov. and gordonia spumicola sp. nov. isolated from sludge of a wastewater treatment plant.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004225
Ou, C. Y., Vu, T., Grunwald, J. T., Toledano, M., Zimak, J., Toosky, M., Shen, B., Zell, J. A., Gratton, E., Abram, T. J., & Zhao, W. (2019). An ultrasensitive test for profiling circulating tumor DNA using integrated comprehensive droplet digital detection.
Lab on a Chip. https://doi.org/10.1039/c8lc01399c
Wang, B., Cheng, H., Qian, W., Zhao, W., Liang, C., Liu, C., Cui, G., Liu, H., & Zhang, L. (2020). Comparative genome analysis and mining of secondary metabolites of paenibacillus polymyxa.
Genes and Genetic Systems. https://doi.org/10.1266/ggs.19-00053
Vaneechoutte, M., Guschin, A., Van Simaey, L., Gansemans, Y., Van Nieuwerburgh, F., & Cools, P. (2019). Emended description of Gardnerella vaginalis and description of gardnerella leopoldii sp. Nov., gardnerella piotii sp. nov. and Gardnerella swidsinskii sp. nov., with delineation of 13 genomic species within the genus Gardnerella.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.003200
Koziaeva, V. V., Rusakova, S. A., Slobodova, N. V., Uzun, M., Kolganova, T. V., Skryabin, K. G., & Grouzdev, D. S. (2019). Magnetospirillum kuznetsovii sp. nov., a novel magnetotactic bacterium isolated from a lake in the Moscow region.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.003408
Doi, H., Uchii, K., Takahara, T., Matsuhashi, S., Yamanaka, H., & Minamoto, T. (2015). Use of droplet digital PCR for estimation of fish abundance and biomass in environmental DNA surveys.
PLoS ONE. https://doi.org/10.1371/journal.pone.0122763
Postel, M., Roosen, A., Laurent-Puig, P., Taly, V., & Wang-Renault, S. F. (2018). Droplet-based digital PCR and next generation sequencing for monitoring circulating tumor DNA: a cancer diagnostic perspective.
Expert Review of Molecular Diagnostics. https://doi.org/10.1080/14737159.2018.1400384
Lipun, K., Chantavorakit, T., Mingma, R., & Duangmal, K. (2020). Streptomyces acidicola sp. nov., isolated from a peat swamp forest in Thailand.
Journal of Antibiotics. https://doi.org/10.1038/s41429-020-0294-5
Nouioui, I., Sangal, V., Cortes-Albayay, C., Jando, M., Igual, J. M., Klenk, H. P., Zhang, Y. Q., & Goodfellow, M. (2019). Mycolicibacterium stellerae sp. Nov., a rapidly growing scotochromogenic strain isolated from stellera chamaejasme.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.003644
Yang, S., Che, S. P. Y., Kurywchak, P., Tavormina, J. L., Gansmo, L. B., Correa de Sampaio, P., Tachezy, M., Bockhorn, M., Gebauer, F., Haltom, A. R., Melo, S. A., LeBleu, V. S., & Kalluri, R. (2017). Detection of mutant KRAS and TP53 DNA in circulating exosomes from healthy individuals and patients with pancreatic cancer.
Cancer Biology and Therapy. https://doi.org/10.1080/15384047.2017.1281499
Feng, X., Wang, Y. R., Zou, Q. H., Zhang, J. Y., & Du, Z. J. (2020). Haloflavibacter putidus gen. Nov., sp. nov., isolated from coastal seawater.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004228
Chen, Y. J., Takahashi, C. N., Organick, L., Bee, C., Ang, S. D., Weiss, P., Peck, B., Seelig, G., Ceze, L., & Strauss, K. (2020). Quantifying molecular bias in DNA data storage.
Nature Communications. https://doi.org/10.1038/s41467-020-16958-3
Nugroho, K., Widyajayantie, D., Ishthifaiyyah, S. A., & Apriliani, E. (2021). Pemanfaatan Teknologi Droplet Digital PCR (ddPCR) dalam Kegiatan Analisis Molekuler Tanaman.
JURNAL BIOS LOGOS. https://doi.org/10.35799/jbl.11.1.2021.31101
van der Putten, B. C. L., Matamoros, S., Mende, D. R., Scholl, E. R., & Schultsz, C. (2021). Escherichia ruysiae sp. Nov., a novel gram-stainnegative bacterium, isolated from a faecal sample of an international traveller.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004609
Menschikowski, M., Jandeck, C., Friedemann, M., Nacke, B., Hantsche, S., Tiebel, O., Sukocheva, O., & Hagelgans, A. (2018). Identification of rare levels of methylated tumor DNA fragments using an optimized bias based pre-amplification-digital droplet PCR (OBBPA-ddPCR).
Oncotarget. https://doi.org/10.18632/oncotarget.26315
O’Keefe, C. M., Li, S., & Wang, T. H. J. (2019). Digital DNA Sequence Profiling of Rare Epigenetic Cancer Biomarkers in A Highly Parallelized Microfluidic Platform.
2019 20th International Conference on Solid-State Sensors, Actuators and Microsystems and Eurosensors XXXIII, TRANSDUCERS 2019 and EUROSENSORS XXXIII. https://doi.org/10.1109/TRANSDUCERS.2019.8808784
Travers, A., & Muskhelishvili, G. (2015). DNA structure and function. In
FEBS Journal. https://doi.org/10.1111/febs.13307
Dorsey, P. J., Rubanov, M., Wang, W., & Schulman, R. (2019). Digital Maskless Photolithographic Patterning of DNA-Functionalized Poly(ethylene glycol) Diacrylate Hydrogels with Visible Light Enabling Photodirected Release of Oligonucleotides.
ACS Macro Letters. https://doi.org/10.1021/acsmacrolett.9b00450
Watanabe, M., Higashioka, Y., Kojima, H., & Fukui, M. (2020). Proposal of Desulfosarcina ovata subsp. sediminis subsp. nov., a novel toluene-degrading sulfate-reducing bacterium isolated from tidal flat sediment of Tokyo Bay.
Systematic and Applied Microbiology. https://doi.org/10.1016/j.syapm.2020.126109
Jia, L. J., Zhang, K. S., Tang, K., Meng, J. Y., Zheng, C., & Feng, F. Y. (2020). Methylobacterium crusticola sp. Nov., isolated from biological soil crusts.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004020
Ye, J. J., Liu, S. W., Lu, Q. P., Cheema, M. T., Abbas, M., Sajid, I., Huang, D. L., & Sun, C. H. (2020). Arthrobacter mobilis sp. nov., a novel actinobacterium isolated from Cholistan desert soil.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004431
Challenge, D. B. (2015). DNA digital data storage. In
rspsciencehub.com.
Fose, L., & Mehl, M. (2007). Plugging into students’ digital DNA: Five myths prohibiting proper podcasting pedagogy in the new classroom domain.
MERLOT Journal of Online Learning and Teaching.
Liu, Y., Lai, Q., Du, J., & Shao, Z. (2017). Genetic diversity and population structure of the Bacillus cereus group bacteria from diverse marine environments.
Scientific Reports. https://doi.org/10.1038/s41598-017-00817-1
Imtiaz M, Jain P, Kment V, & Schaubroeck R. (2019). The building blocks telcos need to create their digital and analytics DNA.
McKinsey & Company.
Qin, P., Zou, Y., Dai, Y., Luo, G., Zhang, X., & Xiao, L. (2019). Characterization a novel butyric acid-producing bacterium collinsella aerofaciens subsp. Shenzhenensis subsp. nov.
Microorganisms. https://doi.org/10.3390/microorganisms7030078
Huang, C. H., Chen, C. C., Liou, J. S., Lee, A. Y., Blom, J., Lin, Y. C., Huang, L., & Watanabe, K. (2020). Genome-based reclassification of lactobacillus casei: Emended classification and description of the species lactobacillus zeae.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.003969
Ping, M., Yun-Lin, Z., Jun, L., Jian, G., & Zheng-Gang, X. (2021). Proposal of Lentzea deserti (Okoro et al. 2010) Nouioui et al. 2018 as a later heterotypic synonym of Lentzea atacamensis (Okoro et al. 2010) Nouioui et al. 2018 and an emended description of Lentzea atacamensis.
PLoS ONE. https://doi.org/10.1371/journal.pone.0246533
Akamatsu, H., Koh, Y., Okamoto, I., Fujimoto, D., Bessho, A., Azuma, K., Morita, S., Yamamoto, N., & Nakagawa, K. (2019). Clinical significance of monitoring EGFR mutation in plasma using multiplexed digital PCR in EGFR mutated patients treated with afatinib (West Japan Oncology Group 8114LTR study).
Lung Cancer. https://doi.org/10.1016/j.lungcan.2019.03.021
Yang, J., Han, X., Liu, A., Bai, X., Xu, C., Bao, F., Feng, S., Tao, L., Ma, M., & Peng, Y. (2017). Use of digital droplet PCR to detect Mycobacterium tuberculosis DNA in whole blood-derived DNA samples from patients with pulmonary and extrapulmonary tuberculosis.
Frontiers in Cellular and Infection Microbiology. https://doi.org/10.3389/fcimb.2017.00369
Tang, Z., Choi, G., Nouri, R., & Guan, W. (2019). Nanopore Digital Counting of Amplicons for Ultrasensitive Electronic DNA Detection.
Technical Digest - International Electron Devices Meeting, IEDM. https://doi.org/10.1109/IEDM19573.2019.8993671
Manzari, C., Oranger, A., Fosso, B., Piancone, E., Pesole, G., & D’Erchia, A. M. (2020). Accurate quantification of bacterial abundance in metagenomic DNAs accounting for variable DNA integrity levels.
Microbial Genomics. https://doi.org/10.1099/mgen.0.000417
Menschikowski, M., Jandeck, C., Friedemann, M., Richter, S., Thiem, D., Sönke Lange, B., & Suttorp, M. (2018). Identification and quantification of heterogeneously-methylated DNA fragments using epiallele-sensitive droplet digital polymerase chain reaction (EAST-ddPCR).
Cancer Genomics and Proteomics. https://doi.org/10.21873/cgp.20088
Huang, A., Zhang, X., Zhou, S. L., Cao, Y., Huang, X. W., Fan, J., Yang, X. R., & Zhou, J. (2016). Detecting circulating tumor DNA in hepatocellular carcinoma patients using droplet digital PCR is feasible and reflects intratumoral heterogeneity.
Journal of Cancer. https://doi.org/10.7150/jca.15823
Wu, Z., Liao, R., Sun, X., Zu, D., Liu, W., Tan, H., & Sun, S. (2017). Digital quantification of DNA by mapping polarization degree related with coding gold nanorods.
Applied Optics. https://doi.org/10.1364/ao.56.009301
Safdar, S., Ven, K., Dillen, A., Lammertyn, J., & Spasic, D. (2018). Hacking DNA for DNA-powered digital bioassay using nazymes.
22nd International Conference on Miniaturized Systems for Chemistry and Life Sciences, MicroTAS 2018.
Bhat, S., Herrmann, J., Armishaw, P., Corbisier, P., & Emslie, K. R. (2009). Single molecule detection in nanofluidic digital array enables accurate measurement of DNA copy number.
Analytical and Bioanalytical Chemistry. https://doi.org/10.1007/s00216-009-2729-5
Teo, W. F. A., Srisuk, N., & Duangmal, K. (2020). Amycolatopsis acidicola sp. Nov., isolated from peat swamp forest soil.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.003933
Taly, V., Pekin, D., Benhaim, L., Kotsopoulos, S. K., Corre, D. Le, Li, X., Atochin, I., Link, D. R., Griffiths, A. D., Pallier, K., Blons, H., Bouché, O., Landi, B., Hutchison, J. B., & Laurent-Puig, P. (2013). Multiplex picodroplet digital PCR to detect KRAS mutations in circulating DNA from the plasma of colorectal cancer patients.
Clinical Chemistry. https://doi.org/10.1373/clinchem.2013.206359
Feng, G. Da, Wang, D. D., Yang, S. Z., Li, H. P., & Zhu, H. H. (2017). Genome-based reclassification of Sphingopyxis ummariensis as a later heterotypic synonym of Sphingopyxis terrae, with the descriptions of Sphingopyxis terrae subsp. Terrae subsp. nov. and Sphingopyxis terrae subsp. ummariensis subsp. nov.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.002465
Zhu, G., Ye, X., Dong, Z., Lu, Y. C., Sun, Y., Liu, Y., McCormack, R., Gu, Y., & Liu, X. (2015). Highly sensitive droplet digital PCR method for detection of EGFR-activating mutations in plasma cell-free DNA from patients with advanced non-small cell lung cancer.
Journal of Molecular Diagnostics. https://doi.org/10.1016/j.jmoldx.2015.01.004
Tian, Q., Mu, Y., Xu, Y., Song, Q., Yu, B., Ma, C., Jin, W., & Jin, Q. (2015). Structure and fabrication details of an integrated modularized microfluidic system.
Data in Brief. https://doi.org/10.1016/j.dib.2015.09.036
Demeke, T., Holigroski, M., Eng, M., & Xing, J. (2016). Absolute quantification of genetically engineered traits with droplet digital PCR: Effect of DNA treatments and spiking with non-target DNA.
Food Control. https://doi.org/10.1016/j.foodcont.2016.03.007
Li, X., & Manz, A. (2019). Precise definition of starting time by capillary-based chemical initiation of digital isothermal DNA amplification.
Sensors and Actuators, B: Chemical. https://doi.org/10.1016/j.snb.2019.02.112
Chen, X. J., Wang, Z. Q., Zhou, Z. Y., Zeng, N. Y., Huang, Q. F., Wang, Z. W., Tang, W. L., & Zhou, H. W. (2020). Characterization of peptacetobacter hominis gen. Nov., sp. nov., isolated from human faeces, and proposal for the reclassification of clostridium hiranonis within the genus peptacetobacter.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.003925
Lam, W. K. J., Gai, W., Sun, K., Wong, R. S. M., Chan, R. W. Y., Jiang, P., Chan, N. P. H., Hui, W. W. I., Chan, A. W. H., Szeto, C. C., Ng, S. C., Law, M. F., Chan, K. C. A., Chiu, R. W. K., & Lo, Y. M. D. (2017). DNA of erythroid origin is present in human plasma and informs the types of anemia.
Clinical Chemistry. https://doi.org/10.1373/clinchem.2017.272401
Jeannot, E., Becette, V., Campitelli, M., Calméjane, M. A., Lappartient, E., Ruff, E., Saada, S., Holmes, A., Bellet, D., & Sastre-Garau, X. (2016). Circulating human papillomavirus DNA detected using droplet digital PCR in the serum of patients diagnosed with early stage human papillomavirus-associated invasive carcinoma.
Journal of Pathology: Clinical Research. https://doi.org/10.1002/cjp2.47
Chen, Y. J., Takahashi, C. N., Organick, L., Stewart, K., Ang, S. D., Weiss, P., Peck, B., Seelig, G., Ceze, L., & Strauss, K. (2019). Quantifying Molecular Bias in DNA Data Storage.
BioRxiv. https://doi.org/10.1101/566554
Garrido-Sanz, D., Sansegundo-Lobato, P., Redondo-Nieto, M., Suman, J., Cajthaml, T., Blanco-Romero, E., Martin, M., Uhlik, O., & Rivilla, R. (2020). Analysis of the biodegradative and adaptive potential of the novel polychlorinated biphenyl degrader rhodococcus sp. Way2 revealed by its complete genome sequence.
Microbial Genomics. https://doi.org/10.1099/mgen.0.000363
Komaki, H., Hosoyama, A., Igarashi, Y., & Tamura, T. (2020). Streptomyces lydicamycinicus sp. Nov. and its secondary metabolite biosynthetic gene clusters for polyketide and nonribosomal peptide compounds.
Microorganisms. https://doi.org/10.3390/microorganisms8030370
Liu, Y., Lai, Q., & Shao, Z. (2018). Genome analysis-based reclassification of bacillus weihenstephanensis as a later heterotypic synonym of bacillus mycoides.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.002466
Song, N., Tan, Y., Zhang, L., Luo, W., Guan, Q., Yan, M. Z., Zuo, R., Liu, W., Luo, F. L., & Zhang, X. L. (2018). Detection of circulating Mycobacterium tuberculosis-specific DNA by droplet digital PCR for vaccine evaluation in challenged monkeys and TB diagnosis article.
Emerging Microbes and Infections. https://doi.org/10.1038/s41426-018-0076-3
Ushio, R., Yamamoto, M., Nakashima, K., Watanabe, H., Nagai, K., Shibata, Y., Tashiro, K., Tsukahara, T., Nagakura, H., Horita, N., Sato, T., Shinkai, M., Kudo, M., Ueda, A., & Kaneko, T. (2016). Digital PCR assay detection of circulating Mycobacterium tuberculosis DNA in pulmonary tuberculosis patient plasma.
Tuberculosis. https://doi.org/10.1016/j.tube.2016.04.004
Xiong, L., An, L., Zong, Y., Wang, M., Wang, G., & Li, M. (2020). Luteimonas gilva sp. nov., isolated from farmland soil.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004197
ul Haq, T., & Shah, T. (2020). Algebra-chaos amalgam and DNA transform based multiple digital image encryption.
Journal of Information Security and Applications. https://doi.org/10.1016/j.jisa.2020.102592
Gan, L., Zhang, Y., Tang, R., Liu, B., Wang, S., Hu, M., Li, Z., & Tian, Y. (2019). Genomic characterization of a potentially novel Streptococcus species producing exopolysaccharide.
3 Biotech. https://doi.org/10.1007/s13205-019-1652-5
Erwin, G. S., Heikkinen, J., Halimaa, P., & Haber, C. L. (2020). Streptomyces lasalocidi sp. Nov. (formerly ‘streptomyces lasaliensis’), an actinomycete isolated from soil which produces the polyether antibiotic lasalocid.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004135
Awan, F., Dong, Y., Liu, J., Wang, N., Mushtaq, M. H., Lu, C., & Liu, Y. (2018). Comparative genome analysis provides deep insights into Aeromonas hydrophila taxonomy and virulence-related factors.
BMC Genomics. https://doi.org/10.1186/s12864-018-5100-4
Prabhu, D., & Adimoolam, M. (2011). Bi-serial DNA Encryption Algorithm (BDEA).
ArXiv Preprint ArXiv:1101.2577.
Vitomirov, A., Ramirez-Gaona, M., Mehta, S. R., & Pérez-Santiago, J. (2017). Random shearing as an alternative to digestion for mitochondrial DNA processing in droplet digital PCR.
Mitochondrion. https://doi.org/10.1016/j.mito.2016.11.005
Liou, J. S., Huang, C. H., Ikeyama, N., Lee, A. Y., Chen, I. C., Blom, J., Chen, C. C., Chen, C. H., Lin, Y. C., Hsieh, S. Y., Huang, L., Ohkuma, M., Watanabe, K., & Sakamoto, M. (2020). Prevotella hominis sp. Nov., isolated from human faeces.
International Journal of Systematic and Evolutionary Microbiology. https://doi.org/10.1099/ijsem.0.004342
Baek, I., Lee, K., Goodfellow, M., & Chun, J. (2019). Comparative genomic and phylogenomic analyses clarify relationships within and between bacillus cereus and bacillus thuringiensis: Proposal for the recognition of two bacillus thuringiensis genomovars.
Frontiers in Microbiology. https://doi.org/10.3389/fmicb.2019.01978
Wang, W., Zhang, G., Yang, J., Gu, H., Ding, L., Yu, H., Yu, M., Cui, Q., Ji, X., & Li, M. (2017). Digital gene expression profiling analysis of DNA repair pathways in colon cancer stem population of HT29 cells.
Acta Biochimica et Biophysica Sinica. https://doi.org/10.1093/abbs/gmw119
Zhou, C., Geng, H., & Guo, C. (2018). Design of DNA-based innovative computing system of digital comparison.
Acta Biomaterialia. https://doi.org/10.1016/j.actbio.2018.09.018
Chouhan, D. S., & Mahajan, R. P. (2014). An architectural framework for encryption & generation of digital signature using DNA cryptography.
2014 International Conference on Computing for Sustainable Global Development, INDIACom 2014. https://doi.org/10.1109/IndiaCom.2014.6828061
Rotondo, J. C., Oton-Gonzalez, L., Mazziotta, C., Lanzillotti, C., Iaquinta, M. R., Tognon, M., & Martini, F. (2020). Simultaneous detection and viral DNA load quantification of different human papillomavirus types in clinical specimens by the high analytical droplet digital PCR method.
Frontiers in Microbiology. https://doi.org/10.3389/fmicb.2020.591452




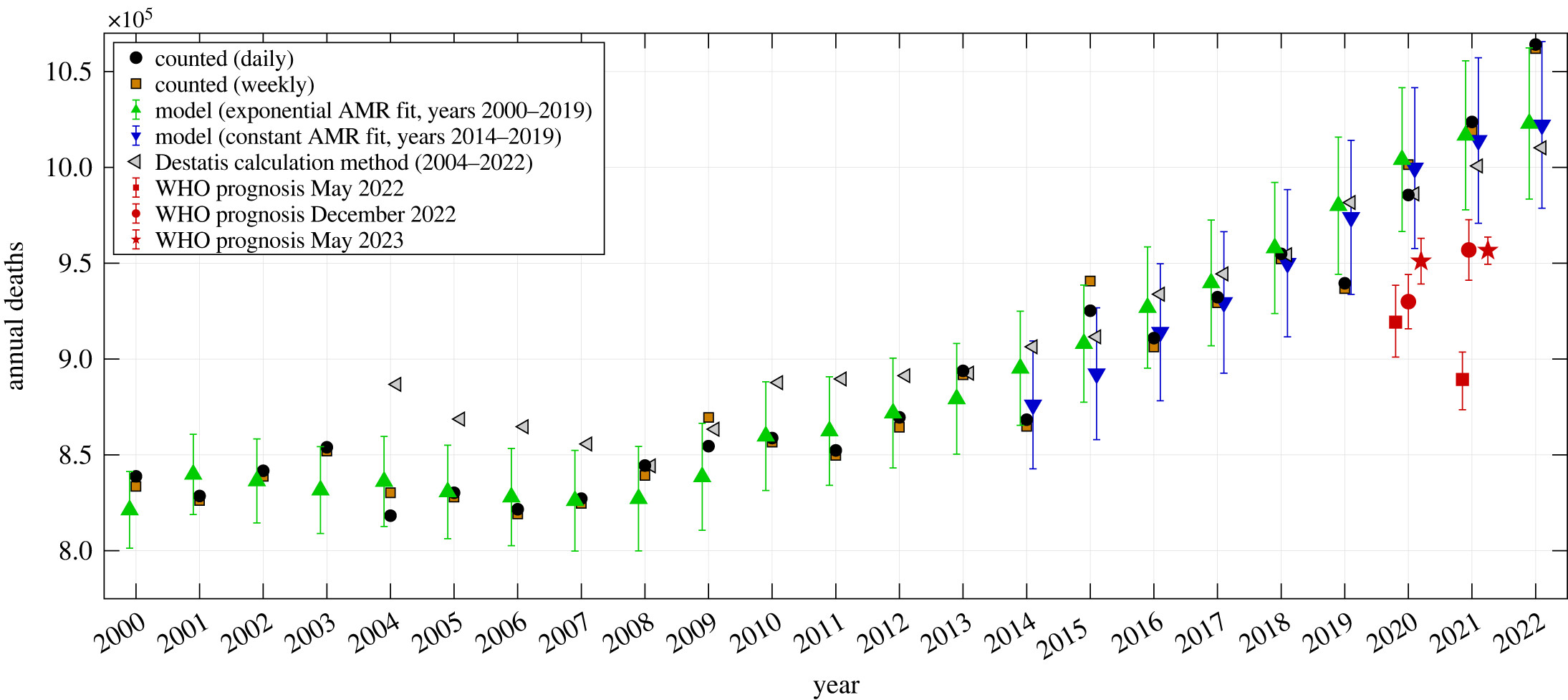

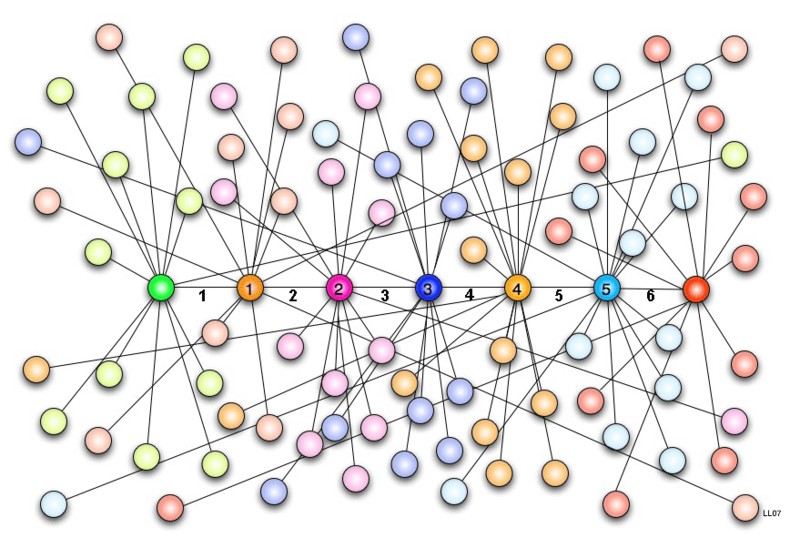

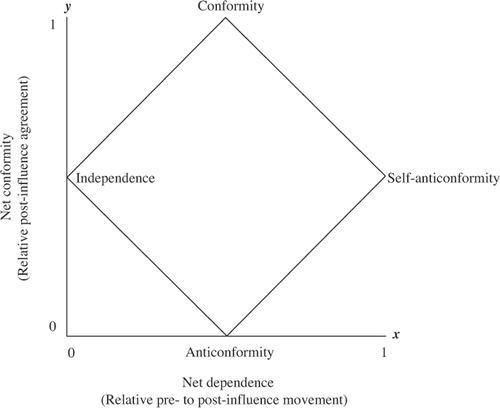

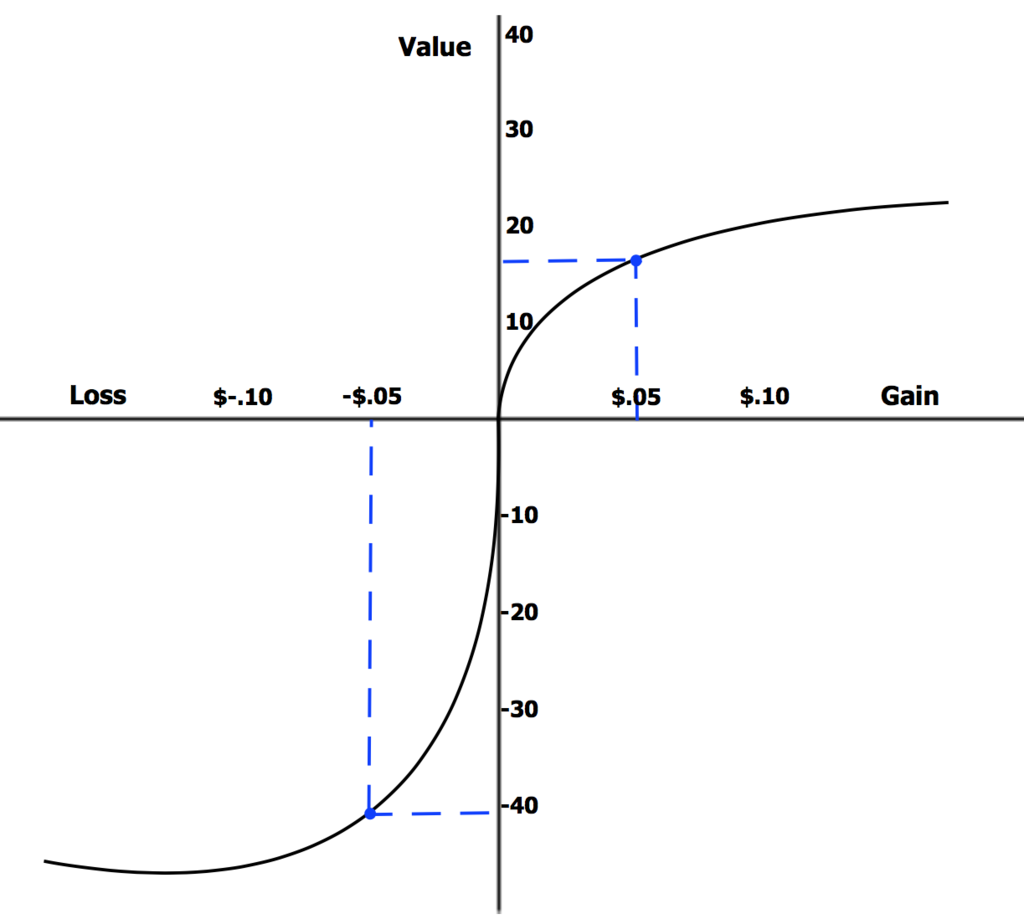







 Wenn man eine Bevölkerung über einen längeren Zeitraum kontrollieren will, muss man ein gewisses Maß an Zustimmung haben. Es ist äußerst schwierig zu erkennen, wie reiner Terrorismus unbegrenzt funktionieren kann. Er kann für eine ziemlich lange Zeit funktionieren, aber ich denke, früher oder später muss man ein Element der Überzeugung einbringen, ein Element, das die Menschen dazu bringt, dem zuzustimmen, was mit ihnen geschieht.
Wenn man eine Bevölkerung über einen längeren Zeitraum kontrollieren will, muss man ein gewisses Maß an Zustimmung haben. Es ist äußerst schwierig zu erkennen, wie reiner Terrorismus unbegrenzt funktionieren kann. Er kann für eine ziemlich lange Zeit funktionieren, aber ich denke, früher oder später muss man ein Element der Überzeugung einbringen, ein Element, das die Menschen dazu bringt, dem zuzustimmen, was mit ihnen geschieht.
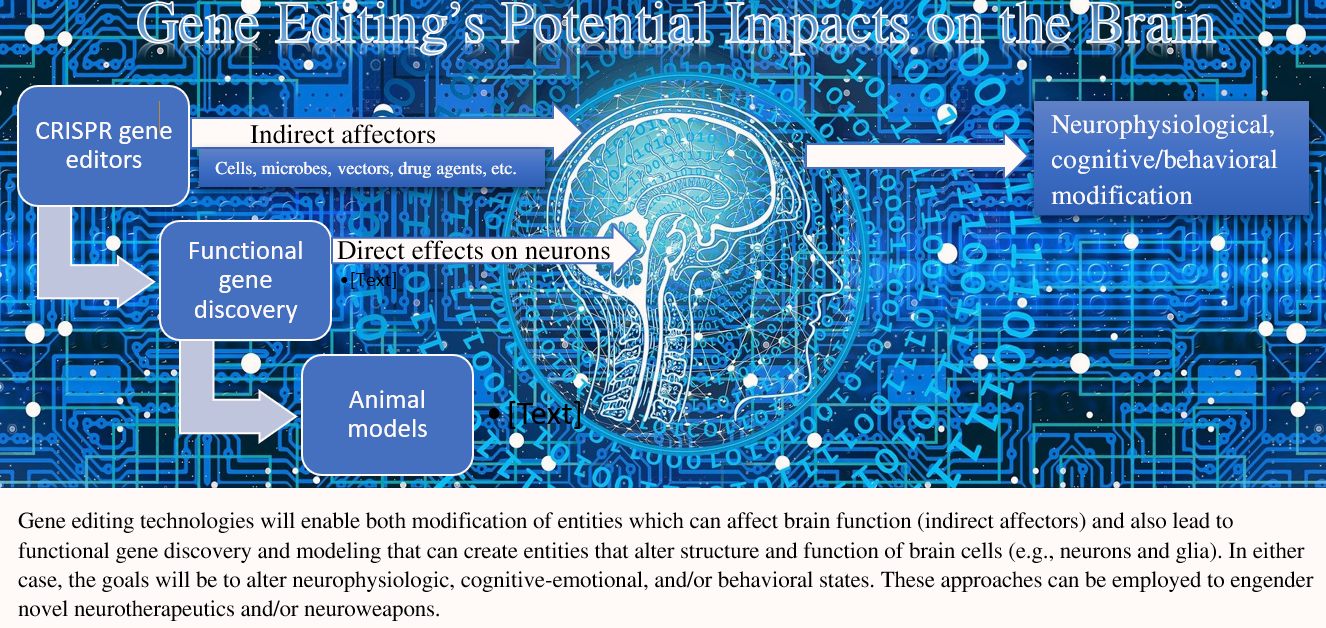



 Die Etymologie der Kybernetik
Die Etymologie der Kybernetik